HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  eXpanda:分子間相互作用ネットワーク可視化ソフトウェアの開発
eXpanda:分子間相互作用ネットワーク可視化ソフトウェアの開発
08.04.10
eXpanda:分子間相互作用ネットワーク可視化ソフトウェアの開発
(08.04.10)
相互作用を自在に描画、細胞内ネットワークを視覚的にとらえて、一段上の理解へ。
1. Negishi, Y., Nakamura, H., Yachie, N., Saito, R. and Tomita, M. (2007) eXpanda: an integrated platform for network analysis and visualization, In Silico Biology, 7, 0013![]()
実験手法の改良や情報処理的な予測手法の確立によって、細胞内のタンパク質同士の相互作用や酵素による基質の触媒作用などを含む、分子間相互作用ネットワークのデータが急速に蓄積されている。この "interactome"と呼ばれるネットワークの集合は今や巨大であり、補助的なツールを使わずに観察することは非常に難しい。また、大量のデータをあつかう必要性があるバイオインフォマティクス解析では、さらにこのような可視化をコンピュータプログラミングによって自動化できなければならない。一方で、プログラミングはどうしてもハードルの高い、労力を必要とする作業である。
&uot そこで根岸・中村らによって開発されたのが、interactomeを様々なウェブデータベースやトポロジー解析と組み合わせて可視化すること ができるソフトウェア eXpanda である。eXpandaはプログラミング言語Perlのライブラリとして実装されており、ライブラリのメソッド群はユーザが記述したコードからいつでも呼 び出すことができるため、Perl言語からのシームレスなアクセスが可能となっている。
eXpandaで利用可能なメソッド数は膨大で、なかでもネットワークに対する解析手法に関しては様々な手法が実装されている。グラフ理 論を元にノードの次数などを計算したり、クリークなどのネットワークモチーフを探し出すことが、わずか数行のプログラムで実行できる。また、様々な形式の データセットと比較したり、既知データセットをウェブ上に点在するデータベースから読み込んで精度検証を行なうことも可能である。
eXpandaはユーザーが構築したネットワークをベクター画像フォーマットであるSVG形式で出力することもできる。それぞれのノードには色や形など13種類の描画要素を、エッジには色や太さなど9種類の描画要素を個別に指定することができる。
このように、さまざまなデータのインポートから解析、グラフィカルな形で可視化を行ってエクスポート、といった一連の作業を、Perlのみを用いて統合的 に行うことができることがeXpandaの強みである。Perlは文字列操作を頻繁に扱う情報生物学において古くから親しまれているプログラミング言語で ある。Perlを利用した経験のある人なら誰でも簡単に利用できるeXpandaは、interactomeに関する解析データを手に入れたい研究者に とって、使いやすくかつ強力な支援ツールである。
例として、eXpandaによって可視化された酵母の分子間相互作用ネットワークを示 す(図)。まず、Cytoscapeから出力されたファイルをインポートし( Step 1. )、グラフィカルな要素のデフォルトの値を適用した( Step 2. )。ついで、各ノードの次数をノードの大きさやラベルの大きさとして可視化し( Step 3.)、KEGGデータベースから読み込んだネットワークデータとの比較を行い、合致した部分グラフを赤く描画するように設定、さらに元々ノードに付加さ れていたSGD entry IDと呼ばれるタンパク質のIDをネットワークAPIを用いて遺伝子名に置換した( Step 4. )。最後に、クリークと呼ばれる完全部分グラフを抽出して緑色で描画した( Step 5. )。このような複雑な処理の実行が、eXpandaを用いると非常に短時間の実装で可能となる。ここで用いられた解析も含め、21個のサンプルスクリプト がソフトウェアのウェブサイトで公開されている。(http://medcd.iab.keio.ac.jp/expanda/sample.html)
eXpanda は発表直後から数多くの注目を集めている。国内の技術誌である日経BP誌のWEB版に取り上げられたことをはじめ、生体内ネットワークの可視化ソフトウェ アとしては最も広く用いられているCytoscapeのWEBページにおいても、"Cytoscapeと連携することで強力な解析が可能なソフトウェア" として紹介されている。現在、世界中の研究者から反応が寄せられており、interactomeに関する研究において非常に強力なヘルパーツールとして、 広く用いられることが期待される。
参考URL
eXpanda http://medcd.iab.keio.ac.jp/expanda/
Cytoscape http://www.cytoscape.org/
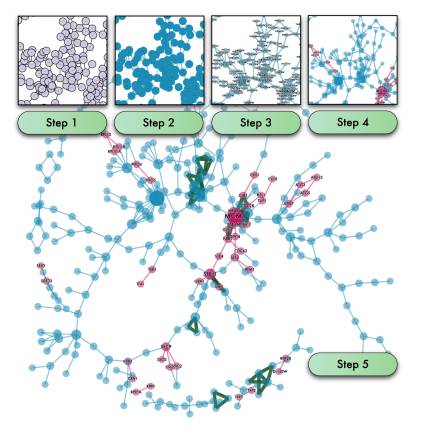
<図の説明>
eXpandaによって可視化された酵母の分子間相互作用ネットワーク。ここに示される5つのネットワークは、解析の過程におけるPerlの構造体の変化を表す。
[ 編集: 小川 雪乃 ]



