HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  SPLITSによるtRNAの網羅的予測と機能解明
SPLITSによるtRNAの網羅的予測と機能解明
08.04.10
SPLITSによるtRNAの網羅的予測と機能解明
(08.04.10)
tRNAを網羅的に予測するソフトウェアSPLITSの開発、そして未知のtRNA発見へ
1. Sugahara, J., Yachie, N., Arakawa, K. and Tomita, M. (2007) In silico screening of archaeal tRNA-encoding genes having multiple introns with bulge-helix-bulge splicing motifs, Rna, 13, 671-81![]()
2. Sugahara, J., Yachie, N., Sekine, Y., Soma, A., Matsui, M., Tomita, M. and Kanai, A. (2006) SPLITS: a new program for predicting split and intron-containing tRNA genes at the genome level, In Silico Biol, 6, 411-8![]()
生物の重要な構成要素であるタンパク質は、原料のアミノ酸を正しい順番でつなげていくことで合成される。この時、messenger RNA上に書かれた遺伝暗号を読み取り、対応したアミノ酸を運んでつなげる役割を担っているのが transfer RNA(tRNA)である。タンパク質を決める遺伝暗号はたった4つの文字ATGCで書かれており、3文字の組み合わせ61種類がアミノ酸を指定している。つまり、生命活動を維持するためには61種類のtRNA遺伝子をゲノム上に持っている必要がある。
tRNAは、配列上の相補的な塩基配列が結合することによってクローバーリーフ構造を形成したり、特徴的な配列を持つことが知られている。そこ で、これらをゲノム配列から探索することでtRNAを予測するtRNAscan-SE、ARAGORNといったソフトウェアが開発、利用されてきた。しか し、古細菌門に属するバクテリア種を中心として必須tRNA遺伝子の多くは未同定であった。
そこで菅原らは、これまでの予測ソフトウェ アでは検出できなかった(1)イントロンを含むtRNA遺伝子(intron-containing tRNA)、(2)二つの遺伝子から合成されるtRNA(Split tRNA)などの、これまでに知られているtRNAとしての特徴を著しく欠く遺伝子も検出することのできる新しいソフトウェアSPLITSを開発した。 SPLITSは、intron-containing tRNAのイントロン、およびSplit tRNAのスプライシングサイトが共通して形成する「Bulge-Helix-Bulge構造モチーフ」をゲノム配列から予測することでtRNA遺伝子を 検出する、ユニークなソフトウェアである。
SPLITSを用いて33種の古細菌ゲノムと1種の真核細胞ゲノム(原始紅藻シゾン)を解析した結果、なんと67もの新しいtRNA遺伝子が発見され、しかもそのうち42の候補が各生物種で未発見であったmissing-tRNAの穴を埋める候補であった。
新しく予測されたtRNA遺伝子はいずれもイントロンを含むものであり、これらのtRNA遺伝子候補の中には単一遺伝子内に3つのイントロンを含むものや (図1)、tRNAの5′-側と3′-側の配列が逆さまにコードされており転写後に環状構造を形成するものなど、過去に報告例のない構造を持った興味深い 遺伝子が数多く観察されている。
現在、SPLITSによって予測された新たなtRNA遺伝子について実験的な検証が進められており、既 に一部の候補については発現が確認されているという。また、SPLITSのさらなる開発と応用によって、tRNA分子の合成やtRNA配列の進化の解明に 向けても日々研究が進められており、近い将来にも未知なる展望が開けてゆくだろう。
参考URL
SPLITS: http://splits.iab.keio.ac.jp
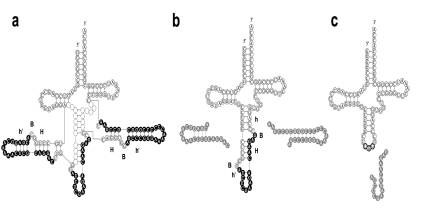
<図の説明>
単一遺伝子内にイントロンを三つ含んだ特殊なtRNAの二次構造図
単 一遺伝子内に三つのイントロンを含んだtRNAの、スプライシングされる過程を模式的に表した。イントロンとして切断される配列を黒丸、エキソンを白丸で 示す(a)。最初に両脇のイントロンが切り取られたのち(b)、真ん中のイントロンが切り取られる(c)という二段階のスプライシング制御が働いていると 考えられる。
[ 編集: 小川 雪乃 ]



