HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  動的/静的ハイブリッドアルゴリズム適用に向けた酵素分類手法の開発
動的/静的ハイブリッドアルゴリズム適用に向けた酵素分類手法の開発
08.02.04
動的/静的ハイブリッドアルゴリズム適用に向けた酵素分類手法の開発
(08.02.04)
メタボロームデータとモデリング手法の融合を果たし、夢の全代謝シミュレーションへ近づく
Ishii, N., Nakayama, Y. and Tomita, M. (2007) Distinguishing enzymes using metabolome data for the hybrid dynamic/static method., Theoretical Biology and Medical Modelling, 4(1), 19
生物の体内では多くの化学反応が起きている。なかでも、生命を維持するために必要な化学反応は「代謝」と呼ばれ、ひとつの細胞で起きる代謝反応は数千にのぼると言われている。このような複雑きわまりないシステムの仕組みを解き明かすには、代謝反応を反応速度式で表現した代謝数理モデルによって、コンピュータ上でシミュレーションを行うことが必須である。
しかし、代謝モデルの作成には通常多数の実験データが必要であり、これは大規模モデルを作る際の大きな障害となっている。そこで、できるだけ実験データを省略しつつ精度の高いモデルを作るための手法として開発されたのが、慶應義塾大学の柚木助教らによる『動的/静的ハイブリッド法』[1]である。これは、代謝系内の酵素を反応速度式で表現すべきもの(動的酵素)と速度式を適用する必要の無いもの(静的酵素)の2種類に分けて計算することで、「静的酵素」の表現に必要なデータの削減を可能とした画期的な手法である。ところが、実際に「動的/静的ハイブリッド法」によって代謝モデルを作成するためには、あらかじめ動的・静的酵素を一定の基準で分類しておかなければならない、という問題が残されていた。
この問題を解決したのが、石井博士らによって開発された判別手法である。これは、細胞内の代謝物質濃度の時間的変化と少数の酵素に関する反応速度式データのみを用い、遺伝的アルゴリズムと呼ばれる最適化計算法によって動的/静的酵素を分類する手法である。
既存の大腸菌[2]および酵母[3]の代謝モデルについて、この手法を用いて最適な動的/静的酵素の組み合わせを見いだし、ハイブリッドモデル(動的/静的ハイブリッド法を用いて構築されたモデル)に変換したところ、元のモデルと比べて十分な精度を得ることができた。更に、代謝物質濃度の測定値にある程度の誤差が含まれていても動的/静的酵素の分類にはほとんど問題が無く、この手法が実際の測定データに対して適用可能であることも確認された。わずかな代謝物質濃度データだけで、全代謝モデル構築に必要なコストの削減を実現したのである。
さて、多数の代謝物質濃度の定量はかつては非常に大変な作業であった。現在は、IABで開発されたキャピラリ電気泳動質量分析装置(CE-MS)を用いることで、メタボローム測定(代謝物質の網羅的測定)が可能となっている。今回開発された手法は、メタボローム測定技術の存在があってこそ、発想できたものである。
石井博士らによる研究は、インフォマティクス研究者が開発した『動的/静的ハイブリッド法』と、実験科学者の開発した『メタボローム測定技術』を結びつけるもので、『情報科学と実験生物学の融合』を目指すIABならではのユニークなものである。夢は、細胞内の全ての代謝をシミュレーションできる『全代謝モデル』の構築。大規模なモデルを効率的に作成できる動的/静的ハイブリッド法の実用化の目処が立ったことで、一歩、この夢に近づいたと言えるだろう。
参考文献:
1. Yugi K, Nakayama Y, Kinoshita A and Tomita M. (2005) Hybrid dynamic/static method for large-scale simulation of metabolism. Theor Biol Med Model. 2:42
2. Chassagnole C, Noisommit-Rizzi N, Schmid JW, Mauch K and Reuss M. (2002) Dynamic modeling of the central carbon metabolism of Escherichia coli. Biotechnol Bioeng 79(1):53-73
3. Hynne F, Dano S and Sorensen PG (2001) Full-scale model of glycolysis in Saccharomyces cerevisiae. Biophys Chem 94(1-2):121-163.
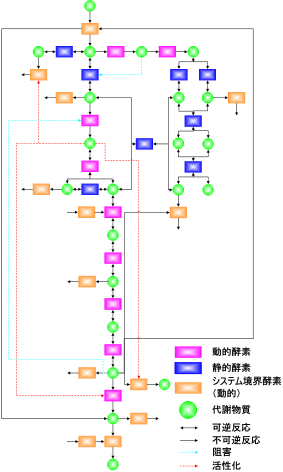
<図の説明>
Chassagnoleらによる大腸菌代謝モデル[2]を題材として静的酵素を判別した結果。システム境界酵素以外の17個の酵素のうち、8個の酵素が「静的酵素」としてモデル化可能と判定された。これらの酵素については反応速度式のデータを省略できる。



