HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  ゲノムを再構築する新しい技術『ドミノ倒し法』の開発
ゲノムを再構築する新しい技術『ドミノ倒し法』の開発
08.04.10
ゲノムを再構築する新しい技術『ドミノ倒し法』の開発
(08.04.10)
枯草菌をベクターに用いて、新たな「合成生物学」のパラダイムへ
Itaya, M., Fujita, K., Kuroki, A., Tsuge, K. Bottom-up genome assembly using the Bacillus subtilis genome vector. Nature methods. 5(1) 41-43.![]()
![]()
ゲノム全塩基配列が迅速に決定される現代、ゲノム中に特定の遺伝子(群)の有無については比較的容易に予想することができる。その次なる段階として、遺伝子群がどのような機能を担っているのかを詳しく調べるために、ゲノムを丸ごと再構築できる実験手法が期待されている。
遺伝子(DNA分子)はそのサイズが比較的小さく、溶液中で生化学的な取り扱いが容易である。そのため、個別に取り出して詳しく調べる手法は大腸菌を宿 主とする遺伝子クローニング法により確立されている。これに対して、ゲノムは細胞の生命活動に必要な全ての遺伝子を含む巨大DNA分子である。ゲノムに含 まれる遺伝子は最小の原核生物マイコプラズマの場合でも480個あり、大腸菌や枯草菌では更に一桁多くなる。このように、ゲノムDNAは巨大な高分子であ るため溶液中では物理的にせん断されやすく、完全長の純粋なゲノムDNAを精製することは極めて困難である。その上、現在最も盛んな大腸菌でのクローニン グにはDNAのサイズの限界がある。従って、ゲノムそのものをクローニングした例はほとんど無い。(ごく最近、米国のVenterらがマイコプラズマゲノ ムの全合成に成功した。画期的な報告であるが、同業者の観点から見ると、彼らもDNAサイズの限界には苦労したようである。参考URL http://www.sciencemag.org/cgi/content/abstract/1151721v1)。
この問題を克服するために、板谷教授らは、DNA組み換え能力の高い枯草菌に注目した。そして、小さな遺伝子断片を出発材料にゲノム全体をクローニング、すなわち再構築するための、実用的な方法を開発した。
この手法は、対象とする長大なDNAを完全にカバーする小さな断片を揃えることから始める。具体的には、PCRで取り扱いやすい大きさ(5 kb程度)のDNA断片を大腸菌でクローニングして用意する。この時、PCRのプライマーを上手に設計して、隣り合う断片では約1 kb程度の配列をオーバーラップさせておく。こうすることによって、DNA断片をドミノに見立てると、オーバーラップ配列の部分であたかもドミノ倒しのように遺伝子断片を次々と連結することができる。ドミノ断片を実際に倒す操作は、枯草菌ゲノム中におけるオーバーラップ部分の相同組み換えを利用している。以上の手順により、多数のドミノDNA断片を順々に連続してつなげ、元のゲノムを枯草菌ゲノム中で再構築する方法を、板谷教授らは『ドミノ倒し法』と命名した。
『ドミノ倒し法』を用いることで、 板谷教授らはマウスのミトコンドリアゲノム(16 kb)を4断片から、イネの葉緑体ゲノム(135 kb)を31断片から、世界で初めて再構築することに成功した。この成果は、ミトコンドリア異常に起因する疾病治療や、葉緑体に新たに遺伝子を組み込むことで植物にワクチンや色素などの有用物質を生産させる技術など、新しい取り組みの今後を切り開く大きな推進力となるであろう。
この技術は汎用性が高く、材料となるゲノムDNAがこまぎれになっていても、PCR法により増幅した遺伝子断片から始めているおかげで、どんなゲノムの再構築にも利用できる。ラン藻ゲノムの完全クローニング(Itaya et al., 2005)、マイコプラズマのゲノム移植(Lartigue et al., 2007)とゲノム全合成の実績を取り入れつつ、『ドミノ倒し法』を適切に利用すれば、近い将来有用微生物の育種をゲノムレベルでデザインすることも可能である。その上、任意に設計した塩基配列を持つドミノ断片を(例えば化学合成法で)用意することさえできれば、『ドミノ倒し法』は完全な人工塩基配列を持つゲノムまでをも、現実のものとすることができるのである。
さて、この革新的な新技術にも、長い試行錯誤の歴史がある。板谷教授らが2005年発表した論文(Itaya et al., 2005)では、枯草菌を用いてラン藻ゲノム(約3500 kb)を枯草菌ゲノム中に再構築することに成功している。そのために開発した『尺取虫法』は、100 kb程度の巨大DNAを一度に取り扱えるほど強力である。だが、調製の難しい巨大なゲノムDNA溶液を必要としていたことと、あまりなじみのない枯草菌を宿主としていたので一般化は難しかった。『ドミノ倒し法』の概念と先行研究は1995年にまでさかのぼり(Itaya, 1995)、その後PCR法と枯草菌ゲノムからの取り出し法(Tsuge and Itaya, 2001)を融合させることで、簡便、かつ普遍的な手法として遂に完成した。しかし、論文になるためには塩基配列レベルでの確認が必要な時代になっており、この点をクリアするのに手間がかかったという。今後、再構築したゲノムは再シーケンスして確認することが必須であり、爆発的に進歩している塩基配列読み取り技術をうまく利用することが求められる。
これまで、生物としてのゲノムを一括して取り扱う研究は実現が困難であり、知的想像をかきたてる以外のものではなかった。しかし、最近の2つの報告(Itaya et al., 2006; Lartigue et al., 2007)で実現可能性が具体的に示されて以降、特に合成生物学(Synthetic Biology)において、重要な中核技術と認識されてきている。合成生物学は、生命現象を理解するために生物を「設計」し「合成」することを目指し、近年注目を集めている新たな研究領域である。『ドミノ倒し法』をはじめ技術的なハードの部分が先に出現しており、塩基配列の青写真を書くソフトの部分が今後の大きな課題である。
青写真の延長線上に、生命をゲノムから創る、という目標がある。実現はまだ先のことであろうが、この目標には賞賛と疑問・批判がある。自然界では、DNAが種・属を超えて移動する水平伝播と呼ばれる現象により、バクテリアゲノムは十億年以上にわたって常に再構築されている。自然のゲノムは再構築ができるほど柔らかく柔軟である、という現実に学び、ラボでのゲノム再構築を安全性に気を配りながら進めていきたい。と板谷教授は語った。
関連文献:
Itaya, M., Tsuge, K., Koizumi, M. & Fujita. K. "Combining two genomes in one cell: stable cloning of the Synechocystis PCC6803 genome in the Bacillus subtilis 168 genome."Proc. Natl Acad. Sci. USA. 102, 15971-15976 (2005).
Lartigue C, Glass JI, Alperovich N, Pieper R, Parmar PP, Hutchison CA 3rd, Smith HO, Venter JC. "Genome transplantation in bacteria: changing one species to another."Science. 317, 632-8. (2007)
Itaya, M. "Toward a bacterial genome technology: integration of the Escherichia coli prophage lambda genome into the Bacillus subtilis 168 chromosome."Mol. Gen. Genet. 248, 9-16 (1995).
Tsuge, K. & M. Itaya. "Recombinational transfer of 100-kilobase genomic DNA to plasmid in Bacillus subtilis 168."J. Bacteriol. 183, 5453-5456 (2001).
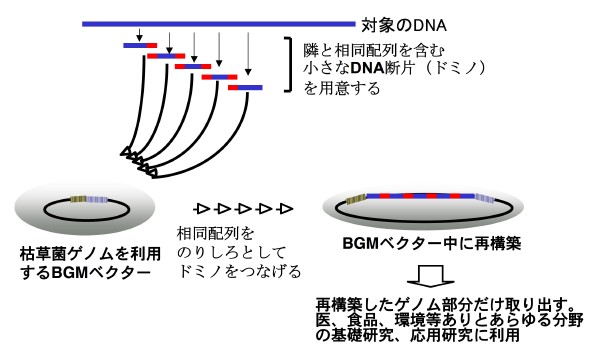
図の説明:
どんなゲノム再構築にも適用可能なドミノ倒し法。重なり合うDNA断片を用 意してつないでゆく。ドミノを倒すのと似ており、巨大なゲノムDNAが再構成できた。ドミノは、既存のゲノムからPCRで得られる。また、完全に人工的に 設計した塩基配列を化学合成して用意することも可能。BGMの由来はBacillus GenoMe。
[ 編集: 小川 雪乃 ]



