HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  温泉に生息する古細菌から最大で3つに分断されているtRNA遺伝子を発見
温泉に生息する古細菌から最大で3つに分断されているtRNA遺伝子を発見
09.07.28
温泉に生息する古細菌から最大で3つに分断されているtRNA遺伝子を発見
(09.07.28)
Tri-split tRNAの発見からゲノム進化の道筋を探る
Fujishima, K., Sugahara, J., Kikuta, K., Hirano, R., Sato, A., Tomita, M. and Kanai, A. Tri-split tRNA is a transfer RNA made from 3 transcripts that provides insight into the evolution of fragmented tRNAs in archaea. , Proceedings of the National Academy of Sciences U. S. A., 106(8), 2683-7. ![]()
![]()
生命の主要な構成成分であるタンパク質は,20種類のアミノ酸が連続的に結合したものである。どのアミノ酸をどの順番で結合するかという情報は遺伝子としてDNAに書かれており、これがDNAからMessenger RNA (mRNA) に転写され、さらにmRNAがリボソームと呼ばれるタンパク質と核酸の複合体に取り込まれてアミノ酸配列に翻訳される。具体的には、リボソーム内でmRNAは3塩基ごとに読まれ、それぞれの3塩基(コドン)に対応するアミノ酸が正確にリボソームまで運搬され、つぎつぎに連結していくことでタンパク質が合成される。このアミノ酸の運搬はリボ核酸であるTransfer RNA(tRNA)が担っていることから、tRNAの進化や原型を調べることで、核酸 (DNAやRNA) からタンパク質への遺伝情報伝達(翻訳系)の生い立ちを明らかにできる可能性がある。
tRNAの起源には諸説あるが、太古のtRNAは短いステムループ構造をとる配列だったのではないかという仮説が現在、幅広く支持されている。近年、系統樹の深い位置から分岐している寄生性古細菌Nanoarchaeum equitansに、tRNAが1つの遺伝子ではなく、2つの遺伝子に分断された状態でコードされているsplit tRNAが発見され、このtRNAが太古のtRNAのなごりである可能性が示唆された。一方でN. equitansは過去に大規模なゲノム縮小が生じていたことが明らかとなったことから、split tRNAはゲノム配列の再編成によって生じた副次的な現象であるという可能性も考えられた。これまでに藤島皓介博士らによって行われたtRNA配列の網羅的系統解析から、split tRNAがイントロン型及び非イントロン型tRNAと同一起源を有するという証拠は得られていたが、tRNA遺伝子の断片化が初期の生命にあったものなのか、あるいは進化の過程で獲得されたものなのかに関しては未だに決着がついていなかった。
こうした状況の中、藤島博士・菅原潤一氏らの研究グループでは、フィリピンの温泉から理化学研究所の伊藤隆先任研究員らによって単離された超好酸好熱性古細菌Caldivirga maquilingensisのゲノムから新たにグリシン、アラニン、グルタミン酸に対応する計10種のsplit tRNA遺伝子を発見した (図1A)。特にグリシンに対応する2つのtRNA (TCC/GCC) は3つの異なる遺伝子から転写された3本の1本鎖RNAが細胞内で部分的に結合し、スプライシングを受けることによって成熟tRNAを作りあげることが示唆され、Tri-split tRNAと名付けられた。
非常に興味深いことに、これらのsplit tRNAはアンチコドン (mRNA上のコドンを認識する部分) に対応するRNA鎖を入れ替えることにより、異なるコドンに対応する成熟tRNAを細胞内で合成していることが示唆された。また、RT-PCR及びPCR産物の塩基配列を決定したところ、実際にこれらのsplit tRNAが転写され、成熟tRNAを合成していることが確認された(図1B、C)。
C. maquilingensisとN. equitansという異なる門に属する古細菌においてsplit tRNAが発見されたことは、短い遺伝子産物を複数組み合わせて機能性のRNA(今回のケースではtRNA)を作る機構が古細菌の共通祖先に備わっていた可能性を示す結果となった。さらに面白いことにC. maquilingensisのsplit tRNAが断片化している位置は近縁種のイントロン介在型tRNAのイントロン配列の挿入位置と完全に一致しており、配列も非常に酷似していた。これはsplit tRNAがイントロン介在型tRNAの祖先である、あるいはその逆である可能性が強く示唆された。
今回の研究で明らかになったTri-split tRNA遺伝子は、複数のピースが集まって初めて意味のあるものを形成することから、藤島博士はこれを「ジグソーパズル」と例え、tRNA以外のRNAにおいても初期の生命システムではジグソーパズル状になっていた可能性を示唆した。もしこのようなジグソーパズルの組み合わせの試行錯誤の中からより長く、より高次の機能を有する機能性non-coding RNAあるいはmRNAが生じてゲノムに固定化されたとすれば,遺伝子重複や水平伝播に代表されるDNAレベルの遺伝子進化とは異なる,RNAレベルでの遺伝子進化の可能性を提示したことになる、と藤島博士は語る。この可能性を探る意味でも古細菌におけるtRNA遺伝子の進化を考えることは非常に重要であり、藤島博士らの今後の研究が期待される。
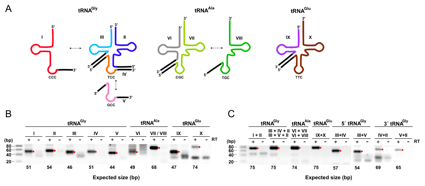
図. 10種のsplit tRNAの組み合わせとRT-PCRによる発現確認
(A) C. maquilingensisにおける10種類のsplit tRNAの異なる組み合わせから作られる6種類の成熟型tRNA。5種類のsplit tRNAGlyから3種類のtRNAGly(CCC/TCC/GCC)が、3種類のsplit tRNAAlaから2種類のtRNAAla(CGC/TGC)、そして2種類のsplit tRNAGluから1種類のtRNAGlu(TTC)が生じている。 エキソン配列はsplit tRNAごとにそれぞれ色分けされている(I, 赤; II, 青; III, 水色; IV, 橙; V, 桃; VI, 灰; VII, 黄緑; VIII, 緑; IX, 紫; X, 茶)。 (B) RT-PCR法による10種類のsplit tRNAの発現確認。(C) 成熟型とプロセス中間型tRNAのRT-PCR法による発現確認。特異的なプライマーにより純粋にRNA産物のみが発現していることを確かめるため、逆転写酵素を含まないサンプルをネガティブコントロールとして用意した(RT ?)。赤い点は予想されたサイズで増幅した産物の位置を示す。



