HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  KEGG Atlasをベースとした多機能ウェブブラウザ「Pathway Projector」の開発
KEGG Atlasをベースとした多機能ウェブブラウザ「Pathway Projector」の開発
10.12.11
KEGG Atlasをベースとした多機能ウェブブラウザ「Pathway Projector」の開発
(10.12.11)
自由に実験データを可視化することができる操作性にすぐれたツールの実現
Kono, N., Arakawa, K., Ogawa, R., Kido, N., Oshita, K., Ikegami, K., Tamaki, S. and Tomita, M. (2009) Pathway Projector: Web-Based Zoomable Pathway Browser Using KEGG Atlas and Google Maps API. PLoS One, 4(11), e7710.![]()
![]()
細胞内の物質は、主にDNA・RNA・タンパク質・代謝物の4階層に大分されており、これらの要素が複雑にからみあって細胞のふるまいを制御している。このような細胞内物質を同定するための高度な技術が近年急速に発展してきているが、それに伴い蓄積される膨大な実験データをどのように解析するかは、現代の生命科学における大きな課題である。そこで注目されるアプローチの一つに、「可視化」がある。例えば、当研究所付近の街並みについて知りたいとしよう。住所のみの文字情報などと比較して、研究所の隣が高校であることや大型スーパーが近くにあるなどといったことを視覚的に示した地図を用いると、相対的な位置関係や周囲の環境などのさまざまな情報を一度に直感的に理解することができる。このように、データの「可視化」は複雑な細胞のふるまいをある程度複雑なまま体系的かつ直感的に理解することを助けることができ、生物学者には必須のツールとなっている。
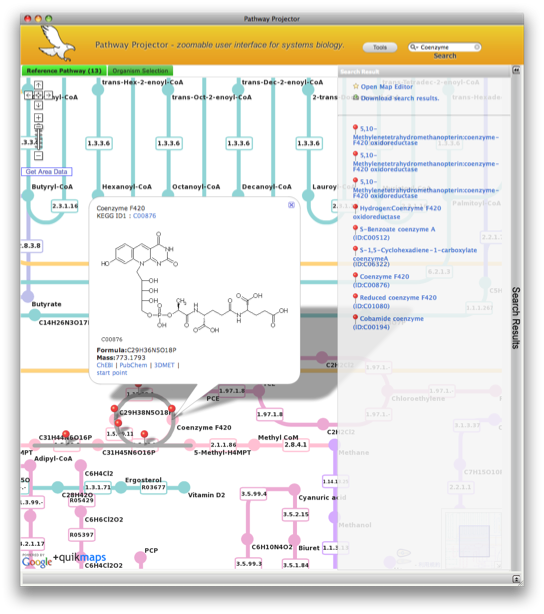
分子生物学で良く用いられている可視化データのデータベースの一つに、Kyoto Encyclopedia of Genes and Genomes(KEGG)が挙げられる。KEGGとは生体内反応経路(パスウェイ)を収集したデータベースであり、酵素による反応の連鎖があたかも電車の路線図のように可視化されている。このようなパスウェイの地図は本来連続的なものであるにもかかわらず、これまでは解糖系やクエン酸回路などといった機能グループごとに分割されていたが、網羅的に細胞を一つのシステムとして理解する重要性から、近年これらを統合したKEGG Atlasなどの大きな地図が提供されはじめてきた。一方で、このような統合パスウェイマップはそれだけで地図として使うのではなく、遺伝子や酵素、代謝産物などの実験データを可視化するプラットフォームとして活用することによって本領を発揮できる。そこで修士課程2年の河野暢明氏は、このKEGG Atlasをベースとして、操作性にすぐれたユーザインタフェースのもとで自由に実験データを可視化することのできるソフトウェア「Pathway Projector」を開発した。
Pathway Projectorの特徴は三つある。一つ目はユーザインタフェースに大規模な地図の操作で定評のあるGoogle Maps APIを採用し、画像遷移を感じさせずにスムーズに拡大・縮小することが可能であることだ。これにより、着目しているパスウェイを中心に、迅速にパスウェイの全体像と詳細を行き来することができる。二つ目は、自身の研究で得られた複数のマイクロアレイ・プロテオーム・メタボロームなどの実験データを同時にパスウェイ上に描画することが可能であるということである。このような異なる階層の細胞内要素を一つずつ可視化するツールは過去にも存在したが、複数の階層を同時に可視化することによって、多角的な視点からシステムとしての細胞を検証・理解することができるようになる。そして三つ目は、興味を持った因子を詳しく調べたい際に、さまざまな検索手法を用いて詳細情報にアクセスできる点である。パスウェイの要素はキーワードだけでなく質量や配列による相同性検索、あるいは二つの要素間の経路などから検索を行うことができ、また生物種間の比較解析なども可能であるという。
Pathway Projectorの開発途中では、多くの困難もあったと河野氏は語る。KEGG Atlasは本来代謝物質の流れを表した地図であり、遺伝子や酵素の情報が個別に描かれていなかったため、これらの情報は手動で注釈付けが必要であった。またGoogle Maps APIによる可視化ではあらかじめ大規模な地図を作っておくことが前提とされており、入力されたデータをもとにした可視化を随時行うことは想定されていなかった。通常この処理は膨大な時間がかかってしまうため、マルチスレッドの分散処理に加え、元データとなるXML形式のファイルの最適化を行うなど、高度な計算機技術による効率化が必要であったという。しかし、結果として従来は10分以上かかる計算処理を約10秒にまで短縮する劇的な高速化を施すことで、ユーザーが使いやすいように工夫をした。
Pathway Projectorは現段階では、KEGGの代謝マップのみ対応している。将来的には、「生物種ごとのデータベースであるEcoCycやAraCycなどの代謝マップへの対応も行いたい。 また代謝マップのみならず、シグナル伝達や膜輸送関連のマップにも対応したブラウザをつくりたい」と河野氏は展望を語る。網羅的な実験と、コンピュータを用いたその高度な解析が相乗効果を生む事により、システム生物学研究が加速度的に発展していくことを期待したい。
[ 編集: 高根香織 ]



