HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  極小イントロンの大規模な転移現象を古細菌で初めて発見
極小イントロンの大規模な転移現象を古細菌で初めて発見
10.12.11
極小イントロンの大規模な転移現象を古細菌で初めて発見
(10.12.11)
遺伝子をコードしないRNA配列の起源やスプライシング機構の一端を明らかに
Fujishima, K., Sugahara, J., Tomita, M., Kanai, A. (2010) Large-scale tRNA intron transposition in the archaeal order Thermoproteales represents a novel mechanism of intron gain. Mol. Biol. Evol., 27(10), 2233-2243.![]()
![]()
遺伝情報の基本単位である遺伝子は、一度RNAに情報が書き写されたのち、RNAのまま機能する領域やタンパク質として翻訳される領域(エキソン)以外は一連の切断反応によって除去(スプライシング)されてしまう。この除かれる不要な領域はイントロンと呼ばれており、タンパク質によって切断されるタイプ、タンパク質とRNAの複合体であるスプライソソームによって切り出されるタイプ、そしてリボザイムによって自己スプライシングするタイプの3種類に大きく分けられる。一般的に知られる真核生物の遺伝子が持つイントロンはこのうちのスプライソソームによって切り出されるタイプのイントロンであるが、その他のイントロンの多様性や役割も注目されてきている。
このような多様なスプライシングの例として、真核生物と古細菌のtRNA遺伝子にはタンパク質によって切断されるタイプのイントロンが見つかっている。面白いことに、その切断酵素であるスプライシングエンドヌクレアーゼも進化的に共に保存されていることから、tRNAイントロンは真核生物と古細菌の共通祖先がすでに有していた起源的に古いタイプのイントロンであることが示唆されている。一方で、その起源や獲得経路に関する知見は現在もほとんどわかっていない。
そこで藤島皓介博士と博士課程2年の菅原潤一氏は、古細菌の中でもtRNAイントロンが多数見つかっているサーモプロテウス目に着目した。彼らの先行研究から、サーモプロテウス目は近縁の古細菌目に比べて複数のイントロンを介在するtRNA遺伝子を多く持つことが確認され、中には最大で3つのイントロンを介在するようなtRNA遺伝子も存在することが明らかとなっている。したがって、急激なtRNAイントロンの増加が見受けられるサーモプロテウス目はtRNAイントロンの進化研究に非常に適していることから、そのtRNA遺伝子やイントロン配列を比較することでイントロンの獲得経路や進化的背景を明らかにしようと考えた。
このため、藤島博士らはまずサーモプロテウス目に属する既知7種の古細菌ゲノムから286のtRNAイントロン配列を抽出し、網羅的な配列アライメントを行い、配列の類似性が70%を超えるイントロンの集団を『同一起源を持つイントロン』と定義することで、合計46のイントロングループを発見することができた。興味深いことに、そのうち16グループにおいては酷似するイントロン配列が進化的に離れているtRNA遺伝子間で見つかったことから、大規模なイントロンの転移が過去に生じていたことが強く示唆された。
ではこのイントロン転移はいつ生じたのだろうか?この疑問に応えるため、イントロン配列の保存性を生物種間で比較してみると、6種にわたって幅広く保存されている古いものから、逆に種特異的なものまでが存在することがわかった。結果、tRNAイントロンの転移がサーモプロテウス目の進化上少なくとも7回起きたことがわかった。また転移現象が頻繁に起きている種ほどイントロンの数が増加していることも確認された。興味深いことに他の古細菌のtRNAイントロンでは同様の現象はまだ確認されていない。
tRNAイントロンは16-32塩基と非常に短いことが特徴として挙げられるが、このように極めて短い配列がどこに由来しているのかはいまだ謎である。藤島らはtRNAイントロンの中でも比較的長い配列(24塩基以上)をNCBIのDNAデータベースに対して検索したが、一つとして該当するものが見つからなかった。したがって、これらはトランスポゾンなどの既知の転移因子由来ではなく、環境中のDNA配列がなんらかのメカニズムによってtRNA遺伝子に取り込まれ、その後個体内の他のtRNA遺伝子にも伝播していった可能性が考えられる。
最後に個体内のtRNA遺伝子間でのイントロン伝播を促している要因として、スプライシング機構を活用したRNAレベルでの伝播が考えられる。これは、転移している単位がまさにエンドヌクレアーゼによって切り出される配列そのものであったからであり、また、転移現象がイントロンの増加と密接にカップリングしていることから、トランスポゾンの転移機構である"カット&ペースト"よりもレトロトランスポゾンのようにRNAレベルで"コピー&ペースト"していると考えた方が腑に落ちる。実際、コンピュータシミュレーションにより、転移可能なtRNAの相手というのはイントロンが挿入された時にバルジ・へリックス・バルジ(BHB)モチーフというRNA2次構造を形成できるかどうかが重要なファクターであることが見出された。
以上の結果は、tRNAイントロンという、古細菌と真核生物が分かれる以前から存在していたであろう遺伝子をコードしないRNA配列の起源やスプライシングの生物学的意義を考える上で非常に面白い発見である。この結果をもとに、将来的にはイントロン転移の分子機構を明らかにしていきたい、と藤島氏は意気込みを語ってくれた。
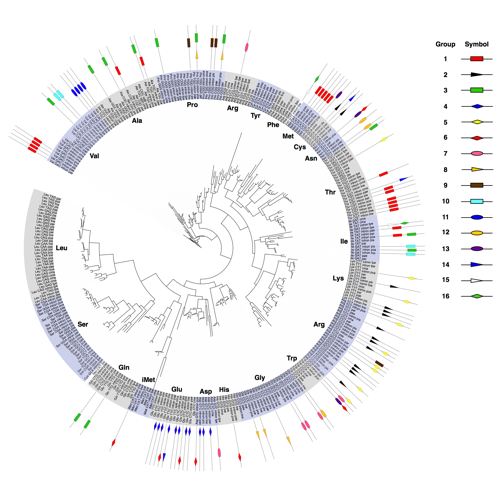
図. tRNA系統樹における転移性イントロンの分布
サーモプロテウス目に属する7種の古細菌から得られた323のtRNA配列を用いて系統樹を作成した。16のグループに属する計113の転移性tRNAイントロン配列は、グループごとに異なる色や形でtRNAにマッピングされている。配列の類似性が70%以上のイントロンの集団を同グループとした。
[ 編集: 高根香織 ]



