HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  バイオインフォマティクスにおけるWebサービスアクセスのためのEMBOSSパッケージ
バイオインフォマティクスにおけるWebサービスアクセスのためのEMBOSSパッケージ
11.11.28
バイオインフォマティクスにおけるWebサービスアクセスのためのEMBOSSパッケージ
(11.11.28)
既存の解析環境にWebサービスを安定的に連携させる新規ツール「KBWS」の開発
Oshita, K., Arakawa, K., and Tomita, M. (2011) KBWS: an EMBOSS associated package for accessing bioinformatics web?services, Source Code for Biology and Medicine, 6:8.![]()
![]()
バイオインフォマティクスの分野では、数千のデータベースや約1500もの解析ツールがインターネット上で公開されており、研究者はそれらを自由に利用することが可能である。このようなWebサービスを活用することで、研究者は煩雑なツールのインストールやメンテナンスを意識することなく、計算資源が豊富なサーバを用いて効率よく解析を行うことができる。
生物学の実験では、遠心機やPCRやDNAシーケンサーなどの装置とさまざまな試薬を組み合わせて一連の研究を行うが、その一連の手順を記録したものが一般的に実験の「プロトコル」と呼ばれる。バイオインフォマティクス研究も同様にさまざまなツールやデータベースを組み合わせて行われるが、このような一連の流れの記述は「ワークフロー」と呼ばれる。このようなワークフローを構築するためにはあるツールの出力結果が他のツールの入力データとして適切な形式である必要があり、そのためにツール間の相互運用性を高めることが近年バイオインフォマティクスソフトウェア開発における中心的課題の一つと位置づけられている。このために、Webサービスにおいては、ユーザの目的に合致したサービスを効率よく発見するための仕組みの開発や、入出力形式の統一化などが国際的な連携の上で進められている。
一方で、現状ではバイオインフォマティクス研究における一連の解析が全てWebサービスのみを用いて行えるわけではなく、実際にはこれらのサービスを如何にしてローカルな計算機環境と連携させていくかが重要となる。そこで、政策・メディア研究科修士課程の大下氏らは、数百のツールを内包し、非常に多くの研究者に使われているバイオインフォマティクスパッケージであるEuropean Molecular Biology Open Software Suite (EMBOSS)に着目した。配列解析用UNIXコマンドラインツール群であるEMBOSSは既に非常に利用者が多く、ほとんど全てのバイオインフォマティクス研究者がローカル環境で使用している。また、EMBOSSは内包しているツールに関して高い相互運用性とサービス探索機能を実現しており、グラフィカルな環境も充実している。そこで、大下氏らは、42個もの主要なWebサービスにアクセスできるEMBOSSの拡張パッケージKeio Bioinformatics Web Service (KBWS)を開発し、既存のバイオインフォマティクス解析環境にすぐにWebサービスの利点を組み込むことを可能にした。
Webサービスには弱点もある。動作安定性がサービス提供者のサーバの状態に左右され、混雑時やメンテナンス時に利用できなかったり、また、指定できるパラメータが類似サービスであっても提供者によって異なるなど、ローカル環境にはない課題も存在する。一方、大下氏らはこの問題に対し、プロキシサーバを用いた中継モデルを採用することで、常時安定してWebサービスを利用可能にした。今後このようなKBWSのフレームワークの下でより多くのツールがサポートされることで、より汎用的かつ多様性のあるソフトウェアになっていくことだろう。
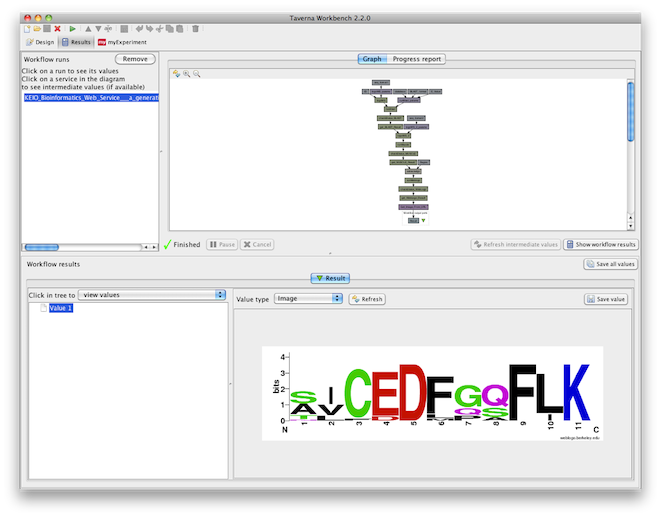
図:KBWSを用いて実際に複数のサーヒスを連携させたワークフローの一例。
FOXP2のシーケンスロゴの画像を作成するワークフローの実行例とその結果を示す。このワークフローは、BLAST Web serviceを用い(kblast)、IDのリストを Uniform Sequence Address (USA) 形式に整形 (sed)、MUSCLEを用いて配列のアラインメント (kmuscle)、アラインメントされた配列の特定の領域を抽出 (extractalign)、抽出された配列を用いてシーケンスロゴを作成する (kweblogo)。
[ 編集:喜久田薫 ]



