HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  物理結合ドメインを考慮したタンパク質複合体の高精度予測
物理結合ドメインを考慮したタンパク質複合体の高精度予測
12.03.01
物理結合ドメインを考慮したタンパク質複合体の高精度予測
(12.03.01)
ドメイン間相互作用を考慮することで劇的に偽陽性を低減
Ozawa, Y., Saito, R., Fujimori, S., Kashima, H., Ishizaka, M., Yanagawa, H., Miyamoto-Sato E., Tomita, M. (2010). Protein complex prediction via verifying and reconstructing the topology of domain-domain interactions. BMC Bioinformatics.11:350.
タンパク質間相互作用 (PPI: Protein-Protein Interactions)データは、効率の良い実験手法の発展により、近年急速に蓄積されてきている。転写や翻訳、代謝、シグナル伝達などの多くの重要 な生命現象には、多様なタンパク質が密に相互作用して形成される複合体が関わっているが、複雑な相互作用データからこのような複合体を予測することができ れば、タンパク質のネットワークがどのように生命現象に関与しているのかを理解することが可能となる。しかし、実際に細胞内に存在する複合体を高精度で抽 出することは難しく、PPIのグラフ表現から複合体を予測する情報科学的手法は複数存在するものの、いずれも偽陽性が多いことが知られている。その理由の 一つは、複合体予測時にタンパク質の同時結合性を考慮していないため、実際には同時に結合できないタンパク質群を複合体として検出しているためである。
そこで小澤陽介博士らは、タンパク質のドメイン間の物理結合(DDI: Domain-Domain Interactions)を考慮した、複合体を形成するタンパク質群の新規予測法を開発した。この予測法では、複合体を構成するタンパク質のリストだけ ではなく、結合のトポロジーと結合を媒介するドメインまで得ることが出来る点が特に画期的である。この物理結合ドメインを考慮することで偽陽性を劇的に低 減させ、さらに複合体予測問題を二値整数計画問題に定式化することで、小澤博士らは効率良く複合体を予測することに成功した。本手法の強みの一つは、高精 度に複合体を予測することで、機能未知タンパク質に対しても、複合体を形成する別のタンパク質から機能を推定することが出来ることである(図)。本手法を 利用することで、233の酵母タンパク質複合体を予測し、そのうち機能未知であった64の複合体のタンパク質の機能決定に成功した。
さ らに、本手法をデータベースに登録されている酵母のDDIデータを用いて検証したところ、MCL (Markov Cluster Algorithm)等の従来法より、約2倍の精度で候補を取得することに成功した。多くの生物種で実験データが充実しているPPIに比べてDDIは入手 可能なデータが少なく、DDIを考慮した複合体予測アルゴリズムを構築することは現状非常に困難である。しかし、この予測手法から得られる高精度な複合体 の候補は実験的にタンパク質複合体を検出する上で直接応用することができ、理想的なバイオインフォマティクスの形を体現しているといえる。今後、二値整数 計画モデルを改良することで、より高精度に生体内の複合体を再現するアルゴリズムを完成させたいと小澤氏は語っている。
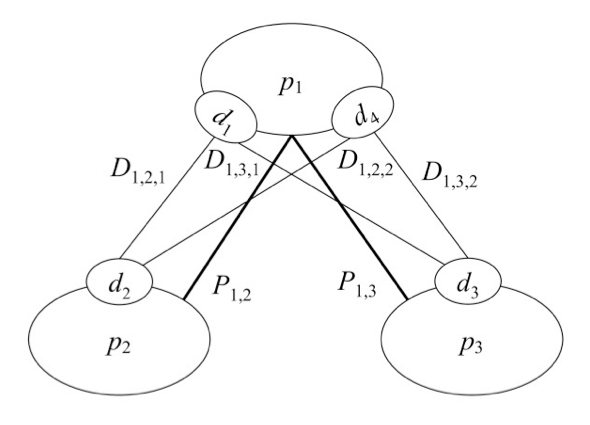
図:予測された複合体の例
ノード(タンパク質)を繋ぐ実線がタンパク質間相互作用(PPI)、点線がドメイン間相互作用(DDI)を示している。下部の例が機能未知タンパク質を 含むタンパク質群から構成される複合体を示している。複合体を構成する別のタンパク質における機能的アノテーションから機能未知タンパク質の機能決定を可 能としている。
[ 編集:喜久田薫 ]



