HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  人工低分子RNAによる細菌の増殖を阻害するシステムの開発
人工低分子RNAによる細菌の増殖を阻害するシステムの開発
12.06.20
人工低分子RNAによる細菌の増殖を阻害するシステムの開発
(12.06.20)
新規人工RNAによる創薬に向けて
Komasa, M., Fujishima, K., Hiraoka, K., Shinhara, A., Lee, B.-S., Tomita, M. and Kanai, A. (2011) A screening system for artificial small RNAs that inhibit the growth of Escherichia coli. The Journal of Biochemistry. 150(3): 289-294.
21世紀に入ってから、数多くの低分子RNAがさまざまな生物種で見つかっている。本研究所のRNA研究グループからも、非常によく研究されている大腸菌 においてでさえも、考えられていたよりずっと多くの低分子RNAが細胞内に存在していると報告がされたばかりである(1)。 このような低分子RNAはタンパク質に翻訳されることなくRNAのままで働くものが多く、そのうちのいくつかは大腸菌の増殖にしたがってその発現量が変化 することが知られている。そこで小正瑞季氏らは、人工的に作成した様々な配列をもつ低分子RNAを細胞内に誘導すれば、その中には、大腸菌の増殖をコント ロールできるようなものが存在するのではないかと考えた。
小正氏らが構築したシステム(図)では、まず、ランダムな60塩基の塩基配列を含んだインサート領域を発現するようなプラスミドライブラリを作成 し、ランダム配列の上流からは翻訳への目印となるリボソームの結合領域を除去して、さらに読み枠をずらした複数の翻訳停止コドンを導入した。これらの処置 により、人工低分子RNAからタンパク質への翻訳を抑制している。次に、この低分子RNA発現ライブラリを使用して、大腸菌の増殖に影響をもたらすクロー ンのスクリーニングを行った。低分子RNAの発現誘導剤であるIPTG(Isopropyl β-D-1-thiogalactopyranoside)の有無で増殖に差があるものが目的のRNAということになる。ここで目的の低分子RNAを効率 よく取得するために、IPTGの有無において次なる連続した2段階のスクリーニングを行なった。すなわち、(i) IPTGを含んだプレート上での大腸菌コロニーの大きさによる選別を行なうこと。(ii) 96穴のプレートを用いて培地の吸光度 (600 nm)を連続的に比較し、IPTG依存 (低分子RNAの発現依存)に大腸菌の増殖を抑制するプラスミドクローンを同定することである。
その結果、これまでに80種以上もの、さまざまなパターンで大腸菌の増殖を抑制する低分子RNAが選別された (図B)。一方、得られた配列を解析しても、増殖の抑制のメカニズムに至る共通性はまだ見えてきていない。将来的にさらに多くの配列と増殖のパターンを比 較し、共通ルールを導きだすことが出来たならば、このような手法は病原性細菌の増殖阻害などへの応用が期待される。小正氏らの研究の発展により、薬として 人工RNAが利用される日も全くの夢物語ではない。
(1) Shinhara, A., Matsui, M., Hiraoka, K., Nomura, W., Hirano, R., Nakahigashi, K., Tomita, M., Mori, H., Kanai, A. (2011) Deep sequencing reveals as-yet-undiscovered small RNAs in Escherichia coli. BMC Genomics, 12, 428.
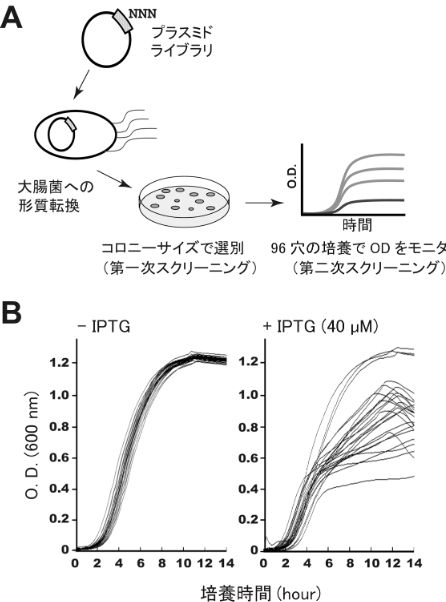
図:大腸菌の増殖を抑制する人工低分子RNAスクリーニング系の開発
(A) 大腸菌の増殖を抑制するような人工低分子RNAのスクリーニング系の概略。 (B) 人工低分子RNAの発現誘導が無し(-IPTG)と有り(+IPTG)の条件下における大腸菌の増殖曲線24例。様々な段階で増殖の抑制を起こすクローン が得られていることが分かる。
[ 編集:喜久田薫 ]



