HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  OryzaPG-DB:ショットガンプロテオゲノミクスに基づくイネプロテオームデータベース
OryzaPG-DB:ショットガンプロテオゲノミクスに基づくイネプロテオームデータベース
12.06.20
OryzaPG-DB:ショットガンプロテオゲノミクスに基づくイネプロテオームデータベース
(12.06.20)
イネにおいて初となるプロテオゲノミクス式データベースの開発 イネにおいて初となるプロテオゲノミクス式データベース
Helmy, M., Tomita, M., Ishihama, Y. (2011) OryzaPG-DB: rice proteome database based on shotgun proteogenomics. BMC Plant Biol., 11, 13.
2012年現在、ヒトをはじめとする数千の生物のゲノム情報が公開されているが、新型シークエンサーの登場により、ゲノム解読の勢いは年々加速している。 ゲノムプロジェクトのねらいは生命の設計図であるDNAの暗号を読み解くことであるが、ゲノム配列そのものは単なるA, T ,G, Cの4文字の羅列であり、実際にはその中のどこに、どういう遺伝子が存在しているのか、を明らかにする作業である「アノテーション」が不可欠だ。従来この アノテーションには既存の知見や、コンピュータ解析、そしてmRNAに発現した遺伝子の情報がもちいられてきた。だが、遺伝子の情報は最終的にはタンパク 質になるのに対し、mRNAはあくまでその中間段階であり、必ずしも精度の良いアノテーションが得られるわけではない。そこで登場したのがプロテオゲノミ クス(プロテオミクス+ゲノミクス)のアプローチである。これはその名の通り、近年急速に発展しつつある質量分析によって得られたタンパク質の情報をもち いて、ゲノムアノテーションの向上を目指す新しい研究分野である。タンパク質の解析は、従来網羅的に行うことが技術的にも、そしてコスト的にも困難であっ たが、質量分析に基づくショットガンプロテオミクスのアプローチは、高い処理量や有効性、相対的な使いやすさなどから、大規模なプロテミクスを可能にし た。
そこで、当研究所の政策・メディア研究科後期博士課程のHelmy氏らは、イネのゲノムのアノテーショ ン向上を目的としてショットガンプロテオゲノミクス解析を行い、その成果をデータベースOryzaPG‐DBとして公開した。従来の2次元電気泳動に基づ くプロテオームのデータベース、例えば RICE PROTEOME DATABASE(Komatsu et al, Proteomics, 2005)とは異なり、本研究では、未分化イネ培養細胞から抽出しトリプシン処理されたプロテオームを、複合イオントラップ/オービトラップ質量分析計を 用いた27回にわたるnanoLC-MS/MS解析によって、精密にペプチドとして測定している(図1A)。測定されたペプチドは、その生成物イオンスペ クトルをミシガン州立大学(MSU)が提供しているタンパク質・cDNA・転写物・ゲノムのデータベースに対して検索することにより、イネゲノムにマッピ ングした(図1B)。OryzaPG‐DBはこの情報を用いてアノテーションが更新された3,182の遺伝子と5,034のタンパク質、その元となる 15,121の非冗長ペプチド配列、さらに、対応するタンパク質、cDNA、mRNAとゲノムの配列を公開している。さらに、このプロテオゲノミクス解析 によって明らかになった新規の166本のペプチド配列により、これまで見つかっていなかった発現領域を新たに40箇所発見することができた。
OryzaPG-DBは、ペプチドベースの発現プロファイルを対応するゲノム情報や新規アノテーションを含めて提供する、世界初のイネプロテオゲノミクス データベースである。本データベースの利用者は、染色体・遺伝子・タンパク質・cDNAそして転写産物ごとにデータベースを検索することができ、また、更 新されたアノテーションを、PNG形式の可視化情報と共にGFF3形式でダウンロードすることが可能である。さらに、同様のプロテオゲノミクスデータベー スを構築するための汎用的なスキーマを合わせて提供することにより、今後のプロテオゲノミクス研究への貢献も視野に入れられている。
イ ネは最も重要な食用作物の1つであり、全世界のおよそ半分はイネに全面的、または部分的に依存していると推定されている。また、イネは比較的小さなゲノム を持つことから研究に適したモデルであり、今後OryzaPG‐DBはイネを研究している多くの生物学者とバイオインフォマティシャンのための有益な情報 源となることが期待される。
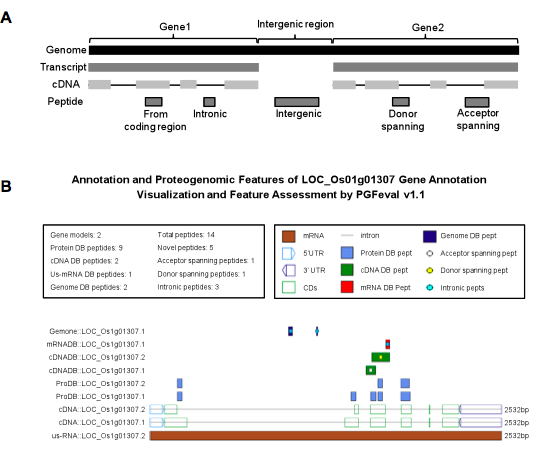
Figure 1. Assessment and visualization of a peptide's genomic novelty. (A) Schematic illustration of peptide clusters. (B) Example of the graphical output of PGFeval, our assessment and visualization tool.
[ 編集:喜久田薫 ]



