HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  環境微生物からtRNA分解に関する新規ルールを発見
環境微生物からtRNA分解に関する新規ルールを発見
12.06.19
環境微生物からtRNA分解に関する新規ルールを発見
(12.06.19)
温泉源泉中の微生物におけるメタトランスクリプトーム解析
Murakami, S., Fujishima, K., Tomita, M., and Kanai, A. (2012). Metatranscriptomic analysis of microbes in an ocean-front deep subsurface hot spring reveals novel small RNAs and type-specific tRNA degradation. Appl Environ Microbiol. 78(4):1015-22.
DNAの遺伝情報はメッセンジャーRNA(mRNA)に転写され、転移RNA(tRNA)を介した翻訳によりタンパク質がつくられる。これは生物共通の仕 組みであり、その中でもtRNAはタンパク質合成に必須な分子として知られている。tRNAはゲノムから転写された後、翻訳されずにRNAのまま働く非 コードRNAの1つであり、さまざまな修飾を経て機能分子となる過程は数多く研究されているものの、成熟後に分解される過程については詳細が明らかになっ ていない。しかし、近年tRNA由来の小分子(tRNAの分解物)が、生体内で機能している可能性を示唆する報告がなされており、tRNAの分解機構の解 明は重要なテーマとなっている。また、これまでの小分子RNA研究は培養可能な種を対象として行われることがほとんどで、難培養性微生物に関する知見は著 しく欠如していた。そこで、政策・メディア研究科修士課程2年の村上慎之介氏らの研究グループは、環境中の難培養性微生物を対象にトランスクリプトーム解 析(メタトランスクリプトーム解析)を行い、tRNAの分解に関して興味深い傾向を見出した。
村上氏らは、山形県鶴岡市の湯野浜温泉源泉中に生息する微生物から抽出した小分子RNA分画を用いてcDNAライブラリーを構築し、次世代シーク エンサーを用いて塩基配列を決定した。4万本を超えるtRNAと4千本を超えるtRNAフラグメントを、図AのようにtRNAの位置毎に切断回数を調査し た。その結果、アンチコドンループとDループと呼ばれる部分で特異的にtRNA分解が起きていることが明らかとなった(図B)。大腸菌では20種中6種の アミノ酸を誘導するtRNAに関してのみアンチコドンループ内でtRNA切断が起こることが知られており、またそれを引き起こす酵素も同定されている。し かし、本研究では20種全てのtRNAの切断様態を観察しており、これまで知られていた6種以外にもアンチコドン付近でのtRNA切断が起こることを報告 している。DループにおけるtRNA分解はバクテリアでは報告がなく新規の知見である。さらに、アンチコドンごとに切断様態を解析したところ、tRNAの 34塩基目と35塩基目の間で切断されやすいtRNAのアンチコドンの1塩基目はAまたはC(プリン塩基)であり、一方でアンチコドンループ以外の位置で 切断されやすいtRNAのアンチコドンの1塩基目はTまたはG(ピリミジン塩基)であることも明らかとなった(図C)。
他にも本論文で は、既知の機能性小分子RNAと相同性の無い新規機能性小分子RNA候補を、湯野浜海岸がボディーボード発祥の地と言われていることからSmall Unique RNA From Yunohama(SURFY:サーフィー)と名付けて報告しているほか、湯野浜温泉源泉中に生息する特殊な古細菌についても報告している。これは、 Archaeal Richmond Mine Acidophilic Nanoorganisms(ARMAN:アーマン)と呼ばれる古細菌の一種で、原始生命の生き残りとして生命誕生の謎を解く手がかりとして重要な生物と 考えられている。
真核生物では、がん細胞などにおいてアンチコドン周辺で分断されたtRNA分子が見つかっているほか、ストレスを受け るとtRNA分解物が多く検出されることなどが知られているが、バクテリアや古細菌におけるtRNA分解機構はあまり理解されていなかった。本研究は、環 境中の難培養性微生物由来の小分子RNAに目を向けたことで、培養可能な微生物からは報告されていなかったtRNA分解に関する新たな特徴を報告した。今 後様々な環境中を調査することでさらなる新規知見の報告に期待すると共に、これらの小分子RNAの生体内における機能理解にも期待したい。
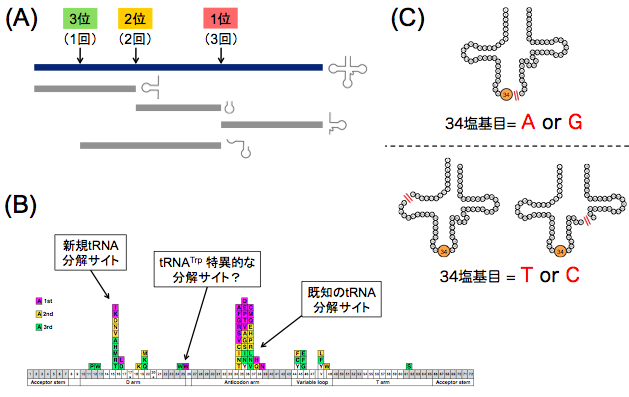
図: (A)tRNA分解の観測法。クローバーリーフ構造を形成できるtRNAにフラグメントをマッピングし、同じアミノ酸を誘導するtRNAごとに切断位置を カウントした。(B)誘導するアミノ酸ごとに(20種のtRNAごとに)上位3箇所の切断位置。ピンクが最もよく切断されていた位置、黄色が2番目、緑色 が3番目となる。(C)アンチコドンごとに切断位置を調べた結果導かれた傾向を示した図。
[ 編集:喜久田薫 ]



