HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  バイオインフォマティクス統合解析プラットフォームEMBOSSとG-languageの融合
バイオインフォマティクス統合解析プラットフォームEMBOSSとG-languageの融合
15.05.03
バイオインフォマティクス統合解析プラットフォームEMBOSSとG-languageの融合
(15.05.03)
IAB発ゲノム解析サービスと外部ソフトウェア間の連携を強化
Itaya, H., Oshita, K., Arakawa, K. and Tomita, M. (2013) GEMBASSY: an EMBOSS Associated Software Package for Comprehensive Genome Analyses. Source Code Biol Med. 8: 17.
バ イオインフォマティクス分野の広がりを象徴するように、さまざまな生物学の分野において数千のソフトウェアやデータベースが専用に開発され、公開されてい る。その結果、現在では多くの解析がそれらいくつものツールを組み合わせることによって可能になってきた。一方で、ソフトウェアが提供されるプラット フォームの違いであったり、入出力ファイル形式の違いなど、ソフトウェア間の相互運用に対しては未だ問題が多く残っており、現代のバイオインフォマティク スにおける根本課題であると考えられている。既にあらゆる解析をするための道具は揃っているか、揃いつつあるにも関わらず、それらを組み合わせて使うため だけに、新たなソフトウェアを作らなくてはならないのではあまりに効率が悪い。そこで、さまざまなソフトウェアの相互運用性を確保する統合解析プラット フォームが開発されてきている。
400以上の配列解析用のUNIXコマンドツールを内包したEuropean Molecular Biology Open Software Suites (EMBOSS) は、最も歴史があり多くの研究者に使用されている代表的な統合解析プラッフォトームの一つだ。EMBOSSに内包されるツールは入出力形式やインタフェイ スが標準化されている他、豊富なドキュメンテーションやユーザインタフェイスが特徴である。これまでに、荒川和晴特任准教授らのグループは、Webサービ スとして提供される多彩なバイオインフォマティクスツールをこのEMBOSSプラットフォームに加えることで、大規模な計算資源を要したり、最新かつ大容 量のデータベースを用いるような解析をも相互運用可能にすることを試みて来た。こうしてEMBOSSの拡張パッケージであるKeio Bioinformatics Web Service (KBWS) が当時政策・メディア研究科修士課程の大下和希氏らによって開発された。今回、このWebサービスによるEMBOSSプラットフォームの拡張をさらに進 め、環境情報学部の板谷英駿氏らは慶應義塾大学先端生命科学研究所で10年以上に渡って開発が続けられている汎用ゲノム解析ソフトウェアG- languageを利用できるEMBOSSの拡張パッケージ「GEMBASSY」を開発した。
G-languageシステムはゲノム解析 を行うための汎用解析環境だが、その中には100種類以上のゲノム解析プログラムが内包されており、その一つ一つが個別に論文として報告されているような 高度かつ独自な解析が多いことが特徴である。特に、ゲノム複製に関わる塩基組成の偏りの解析や、コドンと遺伝子発現量予測、情報量を用いた配列モチーフの 抽出、さまざまなゲノム情報の可視化など、特に原核生物のゲノム解析で強みを発揮する。そこで、板谷氏らは、これらの解析プログラムのうち、特に EMBOSSユーザにとって有益かつ既存のソフトウェアと重複しない機能を52個選び、さらに汎用的にゲノム情報から遺伝子の関連情報を取得するプログラ ムをまとめた計53個のツールをEMBOSSから利用可能にした。この時に、KBWS同様G-language REST/SOAP Webサービスへと接続するように設計することで、これらツールを実行する以外の機能をインストールする必要をなくし、さらにツールのアップデートをする ことなく最新のバージョンをいつでも利用可能にした。配列の入出力などのフォーマットはEMBOSS標準のインタフェイスに統一されているため、ユーザは 実際に動いているプログラムがWebサビスであることを全く意識することなくこれらのツールを使うことができ、他の数百のEMBOSSプログラムと容易に 連携させることができる。EMBOSSはこれまで遺伝子やタンパク質の個別の配列を扱う解析が充実していたが、GEMBASSYを利用することでG- language GAEが得意とするゲノム単位の解析に大きく幅が広がった。EMBOSSという統合解析プラットフォームを通じて、世界中の研究者が慶應義塾大学先端生命 科学研究所で開発されたプログラムやアルゴリズムを活用し、新たな発見に繋がっていくことを期待したい。
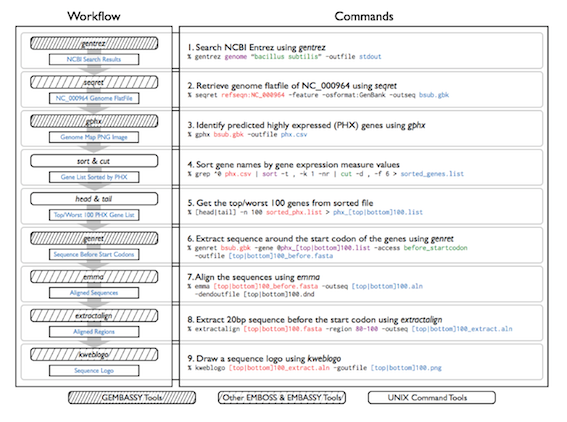
図: GEMBASSYとEMBOSSやUNIXのコマンドラインツールを連携したワークフローの一例。
Bacillus subtilisにおいて予測された高発現遺伝子群と低発現遺伝子群の上流配列保存性の解析の実行例と結果を示す。Bacillus subtilisゲノムの検索と取得を行い (gentrez/seqret)、PHXアルゴリズムによって発現量を計算し(gphx)、発現量の順番でソートを行い (sort/cut)、上位・下位100遺伝子を抽出し (head/tail)、それらの上流配列を取得し (genret)、多重アライメントを行い (emma)、開始コドンの20塩基上流を切り出し (extractalign)、シーケンスロゴを作製する (kweblogo)。
GEMBASSY: http://www.g-language.org/gembassy/
EMBOSS Explorer: http://soap.g-language.org/gembassy/emboss_explorer/
GitHub: http://github.com/ktnyt/GEMBASSY
[ 編集: 川本夏鈴 ]



