HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  50個以上のDNA断片を一度に集積可能な技術を開発
50個以上のDNA断片を一度に集積可能な技術を開発
15.09.20
50個以上のDNA断片を一度に集積可能な技術を開発
(15.09.20)
枯草菌における遺伝子集積法メソッド・OGAB法の革新的改良
Tsuge K, Sato Y, Kobayashi Y, Gondo M, Hasebe M, Togashi T, Tomita M, Itaya M (2015) Method of preparing an equimolar DNA mixture for one-step DNA assembly of over 50 fragments, Sci Rep, 5, 10655発 展と広がりを見せる現代の合成生物学分野においては、自由にデザインされた長鎖DNA(~50 kb)を合成する需要が高まっている。このような新しい配列のDNAを準備するためには、 化学合成したDNAを出発材料にすることが現在の主流である。しかしながら、この手法では数百 bp程度の長さのDNAしか用意できないうえに、一定の頻度で誤りがあるという欠点がある。そのため、オリジナルの長鎖DNA配列を得るためには、短い DNA配列の集積と正しい配列であるかの確認を繰り返し行う面倒な手間が必要である。このコストの高さが原因で、いまだ長鎖DNA合成は研究が進んでいな い。この状況を改善するためには、一度に集積できるDNA断片の数を多くすることで集積と確認の手間を出来る限り減らす必要がある。
2003年に先端生命科学研究所の柘植特任講師らは、OGAB法という枯草菌の形質転換における性質を利用した遺伝子集積技術を開発した。集積の材 料となるDNA断片(OGABブロック)とそれらの集積用プラスミドベクターの各断片の末端に3~4塩基程度の のりしろとなる特異的な配列からなる突出末端を準備し、この突出末端の相補性により、隣り合うOGABブロックを指定して連結する手法である。枯草菌の形 質転換では、大腸菌などと異なり、細胞表層でのDNAの二重鎖切断や、切断サイトからの一本鎖DNAの細胞内への取り込み、そして細胞内での環状化という ユニークな過程を経る。これらの利点を活かした枯草菌による遺伝子集積では、DNA断片が同一方向に繰り返し連結した構造のタンデムリピートDNAが必要 となる。OGAB法では、多数のOGABブロックを試験管内で連結することによりこのタンデムリピート状のプラスミドDNAを調製するが、連結する全ての OGABブロックの分子数が過不足なく揃っていることが重要であった。しかしながら、従来のOGAB法ではOGABブロックの大きさが異なっていたため、 その分子数のバラツキを小さくすることは困難であり、結果的に15個程度のOGABブロックの連結が限界だった。
そこで今回の新しい手法 では、OGABブロックを切り出す際に用いる各種制限酵素の組み合わせのコンピューターシミュレーションにより、可能な限りOGABブロックの長さが等し くなるように設計した。さらに、OGABブロックを組み込んだプラスミドベクターの重量濃度を微量分光光度計によりそれぞれ測定し、すべてのOGABブ ロックプラスミド間で同じになるように調整することで、分子数を厳密に制御することを試みた (図)。これは、従来の方法で必要だったプラスミドベクター部分の除去工程をなくし、OGABブロックプラスミドを混合してまとめた状態で行うという合理 化も同時にもたらした (図)。その結果、各DNA断片の分子数のバラツキの変動係数を従来の20%から7%以下に抑えることが可能となった。
新 OGAB法はこのように効率的にタンデムリピート状プラスミドDNAを準備可能にした。その結果、世界で前例のない、一回の連結操作で50個以上のDNA 断片を指定の向きと順序に連結することが可能な技術が完成し、これまでの酵母を宿主とした遺伝子集積と比較しても倍以上のDNA断片の数を短期間(~5 日)に連結が完了可能である。これにより、設計した50 kb程度の長鎖DNA断片を迅速に構築することが可能となったことで、長鎖DNAを自由にデザインし、有用な機能を生物に持たせるという合成生物学の道具 がまた一つ揃ったと言えるだろう。
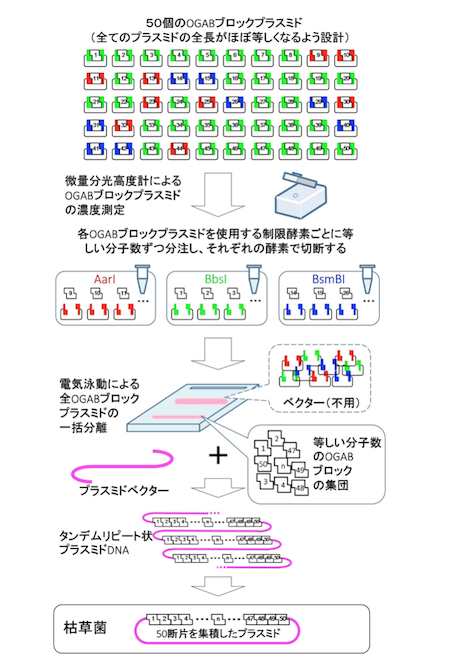
図:今回開発した技術の全体像 この例では、DNA断片をクローンプラスミドから制限酵素で切り出す際に3種類の制限酵素(AarI、BbsI、BsmBI)を用いている。集積対象の配列によっては、より少ない種類の制限酵素を使用することも可能である。
[ 編集: 川本夏鈴 ]



