HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  リボソームプロファイリングによる大腸菌翻訳効率の網羅的解析
リボソームプロファイリングによる大腸菌翻訳効率の網羅的解析
16.02.23
リボソームプロファイリングによる大腸菌翻訳効率の網羅的解析
(16.02.23)
大腸菌遺伝子翻訳効率にまつわるパラドクスを解明
そこで中東憲治特任准教授(当時)らは、リボソームプロファイリングという手法を用い、遺伝子及びコドン毎の翻訳効率の両方を調べることで、上記の問題にアプローチをした。リボソームプロファイリングでは、まず細胞中で翻訳中のリボソームとmRNAの複合体を丸ごと抽出し、これをRNase処理することによって、リボソームとの結合によって守られている、正に翻訳をしている部位だけを取得する。ここで得られた大量の短いmRNA配列断片を、近年急速にその性能が発展している超並列シーケンサーで読み取ることで、細胞中で翻訳されている部位を明らかにすることができる。このようにして得られた膨大なでータを、遺伝子毎の翻訳効率やコドン毎の翻訳効率という観点から比較解析を行った結果、使用頻度の多いコドンの並びによる翻訳効率への影響は確かに存在するが、遺伝子毎の翻訳開始効率の違いによる翻訳効率の方がはるかに大きいことを示した。つまり、個々の遺伝子の翻訳効率の調節にはコドン自体の効率はあまり寄与しないものの、最適な同義コドンの選択はゲノム全体として翻訳装置の効率を上げることには貢献していると考えられ、一見矛盾する先行研究の両方の結果を矛盾無く説明できることが分かった。
超並列シーケンサーの発達と普及により、生物の網羅的かつ定量的な理解が進んできているが、それがリボソームプロファイリングのような新しい手法と組み合わされることによって、我々は今まで見ることができなかった生命現象を解明することが可能となってきている。例えば、リボソームプロファイリングによって、タンパク質合成阻害による抗菌作用を持つマクロライドやクロラムフェニコールなどの抗生物質の詳細な作用機序が近年明らかになってきている。このような幅広い翻訳機構の解明に向けて、これから大腸菌を色々な環境で生育し、遺伝子毎やコドン毎の翻訳効率が遺伝子発現の調節にどのように効いているかを調べていきたいと中東氏は語った。
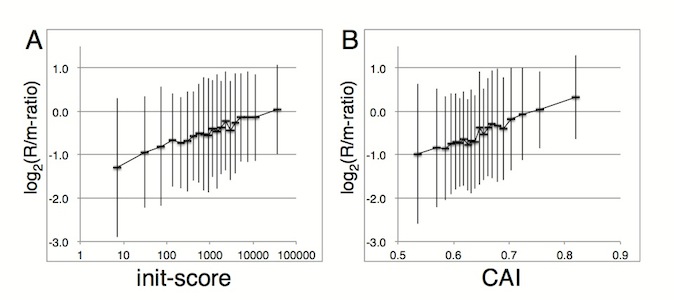
図: mRNAの量に対するリボソームプロファイリングにより得られた配列数の割合と翻訳開始効率及びコドンの並びの関係性。
A及びBの縦軸はmRNAに対してリボソームが結合した割合を示している。Aの横軸はinit-scoreと呼ばれる指標であり,本研究ではmRNAの翻訳開始の効率の良さを示す。Bの横軸は遺伝子のコドンのならびによる翻訳効率を示している。



