HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  微小生物の単一個体由来の超微量DNAによる全ゲノム解析手法を確立
微小生物の単一個体由来の超微量DNAによる全ゲノム解析手法を確立
16.11.18
微小生物の単一個体由来の超微量DNAによる全ゲノム解析手法を確立
(16.11.18)
クマムシの進化をめぐる議論にも大きな進展
Arakawa, K., Yoshida, Y., Tomita, M.(2016) Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 2016 Aug 16;3:160063. doi: 10.1038/sdata.2016.63.
水は生命の源であると同時に、生物もその7割程度は水によって構成されている。私たちを形づくる細胞は水で満たされており、水が溶媒となってさまざまな化学反応が起こり、生命活動に必須であるタンパク質の合成や代謝が行われる。過剰な水分の喪失はすなわち死を意味するが、一部の生物はその常識を覆すように進化した。つまり、周囲の乾燥に伴い自らの水分を完全に失った仮死状態に入り、再びの吸水によって生命活動を再開するのである。地球上に遍在する微小動物クマムシがその一例である。クマムシは脱水に伴い「乾眠」と呼ばれる可逆的な生命活動の停止が可能で、この状態では宇宙空間を含めたさまざまな極限環境に耐性を示す。このような特殊能力に加え、クマムシは独自に緩歩動物門を形成するように、進化分類上も興味深い特性を持つ。
2015年12月、アメリカ ノースキャロライナ大学(UNC)のグループが、クマムシの一種であるHypsibius dujardini (ドゥジャルダンヤマクマムシ)のゲノムを決定したという報告を米国アカデミー紀要に報告した。この論文では、驚くべきことに、そのゲノムの実に17.5%もの遺伝子が水平伝播、つまり他の個体からゲノムが取り込まれることによって獲得されたものだと結論づけた。通常遺伝子は祖先から子孫へと受け継がれる中で、少しずつ変異が蓄積し、選択されることで緩やかに変化を続けている。主に微生物で頻繁に観測される水平伝播は、このような直列の遺伝ではなく、周囲に存在する他の生物が持つ遺伝子を取り込むことによって不連続かつ急激な進化を起こす現象である。H. dujardiniのゲノムで観察されたように、その生物の設計図であるゲノムの実に1/6もの情報が外部から獲得されてしまう現象が実際に起こりうるのであれば、それは生物種というものの自己同一性を根底から問い直さねばならない大発見であるため、この結果は直ぐに大きな驚きを持って各種メディアに受け止められた。
一方で、UNCグループの報告には疑問点も多く存在した。まず、解読されたとされるゲノムのサイズが、他グループによる過去の実験によって推定された値の倍以上となる極めて大きなものであった。また、生物のゲノムを新たに同定する場合には、断片的にシーケンスしたゲノム情報をつなぎあわせるゲノムアセンブリーという工程が必要となる。UNCグループの報告にあったゲノムアセンブリーの統計値は優れておらず、特に、本当に決定されたゲノム情報中にクマムシ以外のゲノムがコンタミネーションしていないのか、という点についてその手法にも疑わしい点があった。クマムシは1mmに満たない微小動物であり、あまりに小さいために通常は数千から数万個体からDNAを抽出することで初めてゲノム解析が可能になる。さらに、飼育系がまだ発展途中である現在、クマムシは無菌状態で飼育されているわけではなく、DNAを抽出するために多数のクマムシを集める過程で、飼育環境中の微生物も一緒に含まれてしまう可能性があるためである。このようにコンタミネーションが含まれてしまうと、何が本当の水平伝播遺伝子なのかを見分けることは事実上困難となる。UNCグループの報告のこのような疑問点はすぐに多数の研究者によってTwitterを始めとするソーシャルネットワーク上で議論され、いくつかのグループが公開された配列データの再解析などにより、UNCグループの報告には大量のコンタミネーションが含まれていることが徐々に明らかとなってきた。
慶應義塾大学先端生命科学研究所の荒川和晴特任准教授のグループは、クマムシの分子生物学的解析時にそのサイズやコンタミネーションが問題になる点を克服すべく、兼ねてより微量解析系の開発を進めており、クマムシ一匹から抽出可能な約50pgのDNAから全ゲノム解析を可能にする手法を確立した。そして、今回その新手法でH. dujardiniの同一株を、絶食させることによってエサとなる生物を排除し、抗生物質処理後に高解像度顕微鏡で観察しながら表面のバクテリアを完全に洗い流した状態の一匹をもちいることによって、コンタミネーションのないゲノムシーケンスを行った。その結果、実にUNCグループのゲノム配列の30%以上が非クマムシ由来のコンタミネーションであることを明らかとした。クマムシは特異な能力を持つ極限環境生物であるが、さすがに積極的に外部のDNAを取り込むような前代未聞のモンスターではなかったのである。
「ピアレビューによる学術論文査読システムは稀に機能しない場合があり、今回のように間違いを含む結果が世にでてしまうことは残念ながらあり得てしまいます。一方、科学は発表後に第三者が検証し追証することで自浄作用を持ち、その時には論文と共になるべく完全な実験データが公開される必要があると思います。」このような流れはオープンサイエンスと呼ばれており、荒川特任准教授はその重要性についても言及した。「近年の論文のオープンアクセス化、データ公開の義務化、そしてソーシャルネットワークによる科学コミュニティ内のオープンかつ迅速な議論が可能になったことなどによって、このオープンサイエンスの流れは主流になりつつあると思いますし、オープンサイエンスは科学を健全なかたちで発展させる上で重要な役割を担いつつあるのではないでしょうか。」と、荒川特任准教授は語った。
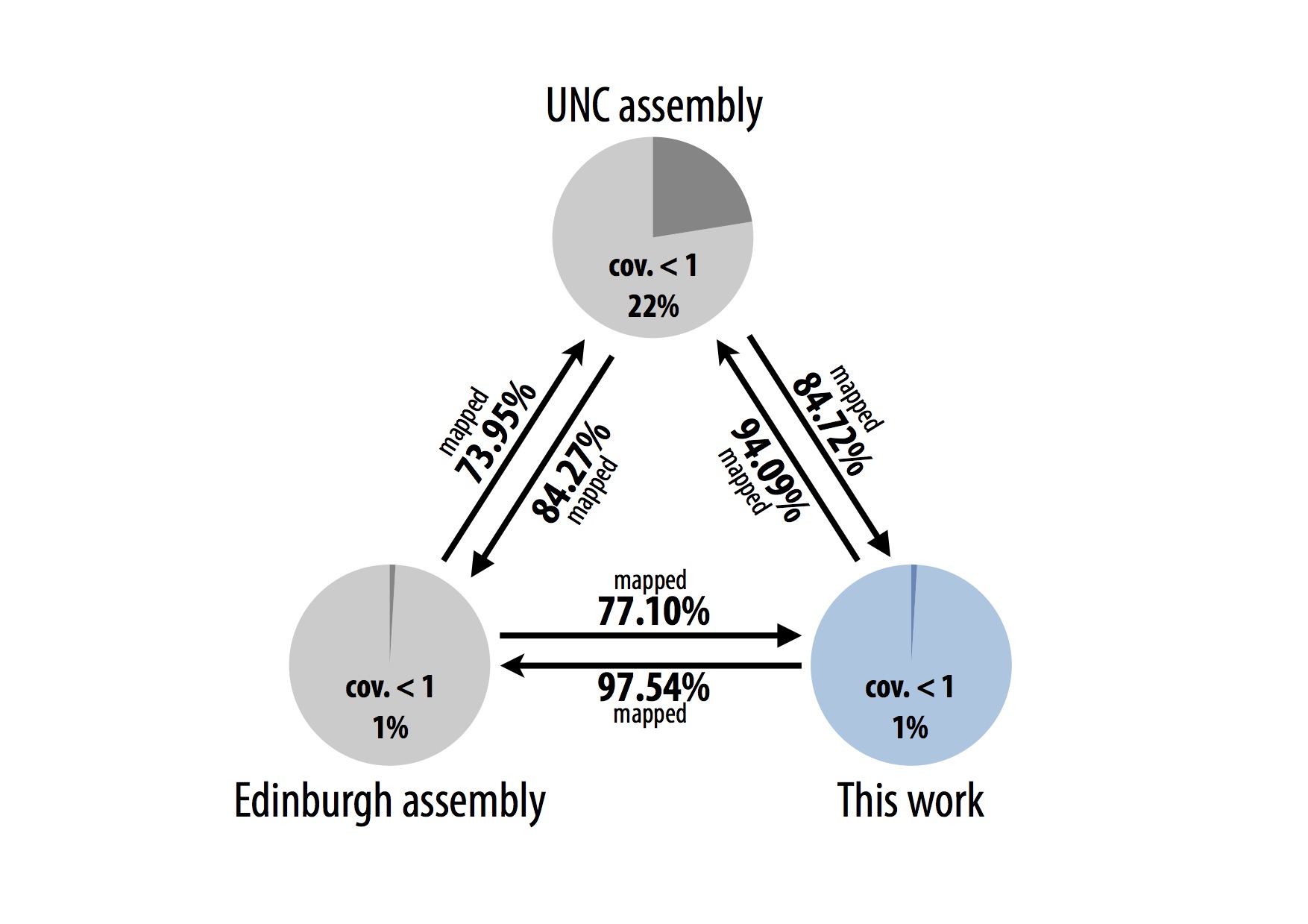
図:本研究と既存手法で得られた H. dujardini の配列比較
今回、超微量シーケンスによって得られた配列のほとんどは、既存の手法を用いて得られた配列中に含まれていた (UNC assemblyに対して94.09%、Edinburgh assemblyに対して97.54%)。一方で、既存の手法で得られた配列のうち、10?25%は本研究では確認ができず、コンタミネーションによるものであることが示唆された。
[References]
Arakawa, K. (2016) No evidence for extensive horizontal gene transfer from the draft genome of a tardigrade. PNAS. 2016 May 31;113(22):E3057. doi: 10.1073/pnas.1602711113.
Arakawa, K., Yoshida, Y., Tomita, M. (2016) Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 2016 Aug 16;3:160063. doi: 10.1038/sdata.2016.63.
[編集:川本 夏鈴]



