HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  細胞内のゲノム挙動情報を活用したコンティグ再配置アルゴリズムを開発
細胞内のゲノム挙動情報を活用したコンティグ再配置アルゴリズムを開発
18.06.16
細胞内のゲノム挙動情報を活用したコンティグ再配置アルゴリズムを開発
(18.06.16)
ドラフトゲノムからゲノム構造の推定が可能に
Kono, N., Tomita, M. and Arakawa, K.(2017) eRP arrangement: a strategy for assembled genomiccontig rearrangement based on replication profiling in bacteria. BMC Genomics.2017 Oct 13;18(1):784. doi: 10.1186/s12864-017-4162-z.
もしも未知の生物と出会ったら、あなたはまず何をするだろうか?写真を撮影し、形態が類似している別の生物と比較をするだろうか?確かにその情報も重要だが、未知の生物を理解するためにぜひ手に入れるべきは、ゲノム配列情報である。ゲノムとは生物が持つ遺伝子情報の総体で、A,T,G,Cの4種の塩基によって構成されている。対象とする生物がどのような遺伝子を持つかを知るためには、ゲノム配列の詳細を知ることが必須である。そして、今ではそれが比較的安価に手に入る。
これまでのバクテリア研究では、主に大腸菌などといったモデル生物が対象とされてきた。しかし近年では非モデル生物、いわば未知の種についても、ゲノム配列の決定とそれに基づく詳細な解析が容易に行われるようになってきている。非モデル生物のゲノム配列解読の敷居が低くなった要因としては、ゲノム配列を解読するシーケンサーの技術革新はもちろんであるが、その解読結果を統合するアセンブラの開発が挙げられる。アセンブラは細かくちぎられた紙を修復するように、細かいDNA断片の配列を読み取り、ゲノム断片のまとまりである「コンティグ」へと統合するツールである。一方で、未だに複数のコンティグをひとつなぎの完全なゲノムDNA配列とするには依然として大きなコストがかかる。そこで、この「コンティグ」に基づいた一歩手前の完成度のゲノムである「ドラフトゲノム」を使って解析が行われる場合が多い。
しかし、「コンティグ」の情報のみでは、ゲノム構造に関する情報を無視してしまう問題がある。コンティグをまたぐような大局的な解析ができないからである。そのため、仮に対象とする非モデル生物について近縁種の情報があったとしても、ゲノムの逆位変異や、遺伝子の配置順などのゲノム構造を把握することができない。特にバクテリアでは、ゲノム構造が細胞内の挙動にも大きく貢献しているため、ゲノム構造を明らかにできなければバクテリアの進化過程について議論することが難しい。
これらの問題を解決するため、慶應義塾大学先端生命科学研究所の河野暢明特任助教(当時)らは、ドラフトゲノム情報からゲノム構造を明らかにできる手法開発に取り組んだ。その結果、生物学的な分子挙動情報に基いて、ゲノム上におけるコンティグ間の位置関係を類推するアルゴリズムeRP arrangement を発表した。環状なバクテリアゲノムでは、通常1つの複製開始点から対称に位置する終結点へと染色体が複製されるが、そのため微生物の集団としては、僅かながら複製開始点付近のDNAの方が終結点付近よりもコピー数が多い。しかも、その量は複製開始点から終結点に向けて徐々に低下する勾配となっている。このようなゲノム中のDNAの量の差は通常無視されているが、今回発表されたeRP arrangementは、このDNA量の勾配(eRP) を利用したものである(Kono, et al., 2014)。アルゴリズムは大きく分けて三段階で構成されており、まず勾配が生まれやすい対数増殖期に敢えてサンプリングしたゲノム(図A) をシーケンス・アセンブルして初期コンティグを作成する(図B)。次に、初期コンティグのなかにあるDNA量勾配をリードのマップ数から算出する(図C)。最後に、その結果で得られたDNA量の勾配が複製開始点・終結点を起点にV字型になる様に編集し、ゲノム上のコンティグ間の位置関係を推定する(図D)。実際にeRP arrangementの効果をテストするため、ゲノム構造に異常がある変異体、つまり、ゲノムの配列は基本的に同一ながらもその位置関係が大きくシャッフルされたものを対象としてeRP arrangementを利用したところ、ゲノム内の長距離関係を直接示すデータが無い状態でも、ドラフトゲノムに隠された勾配情報のみからゲノム構造を高精度で推定できることも確認された。
既存のアセンブリは基本的に塩基配列をベースとしているものしかなく、その部位ごとの「量」に関する情報は見過ごされていた。対して、今回発表されたeRP arrangementは細胞内挙動に基づくDNA量の勾配を利用しており、シンプルながら全く新しいものである。また、低コストで得られるドラフトゲノム情報から、ゲノム構造を理解することが可能になったことは、今後のバクテリア研究における進化の議論に大きく貢献することが予想される。「論文中では最適なサンプリングタイミング、DNA抽出後のライブラリプレップ、必要シーケンス量、解析アルゴリズムなど、eRP arrangementを利用するための詳細も提供しています。モデル・非モデル生物の壁は測定技術が足かせとなっている部分があるので、そこを取り払っていきたいです」と、河野氏は展望している。
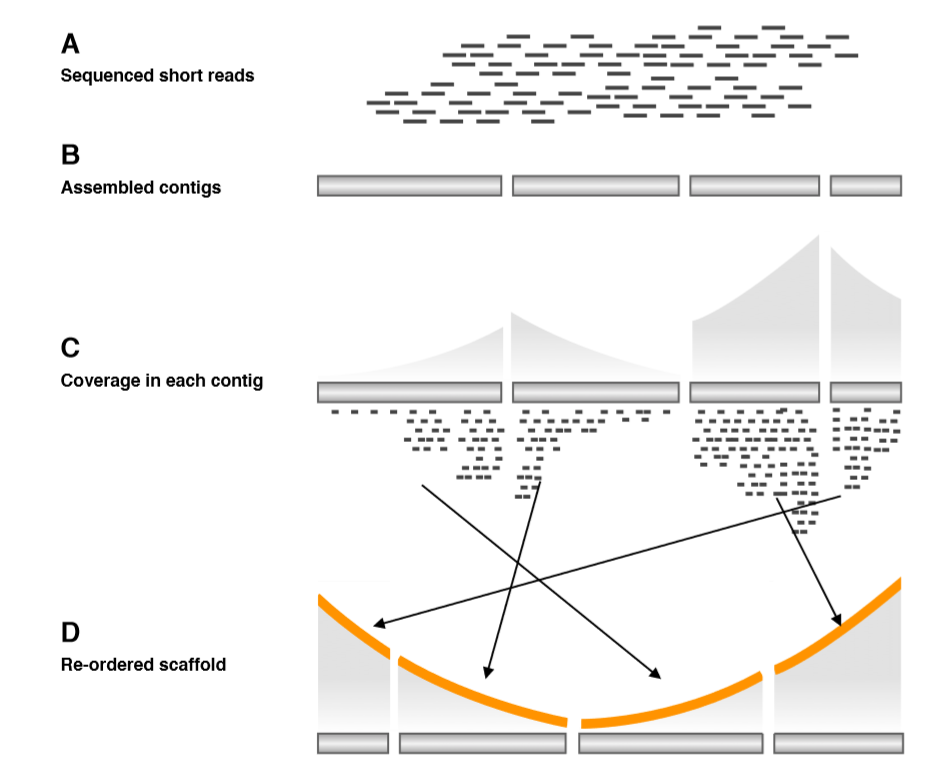
図: eRP arrangementのアルゴリズム
(A) eRP形成のため定常期より手前でゲノムDNAをサンプリングする。(B) シーケンス・アセンブルして初期コンティグを作る。(C) 初期コンティグにリードをマップし、コンティグ毎にDNA量の勾配を計算する。この時サンプリング時期を調整しているためにゲノム全体でDNA量がV字型に偏り、eRPが形成されている。(D) そのためDNA量の勾配がV字型になる様にコンティグを並び替え順番を決定することで、ゲノム構造の情報を得る。
[編集:川本夏鈴]



