HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  周期的な配列を有するゲノム編集関連配列を検出する新規ソフトウェアを開発
周期的な配列を有するゲノム編集関連配列を検出する新規ソフトウェアを開発
19.05.23
周期的な配列を有するゲノム編集関連配列を検出する新規ソフトウェアを開発
(19.05.23)
大規模ゲノムデータからの周期的DNA/タンパク質配列の網羅的捕捉に成功
Mori, H., Evans-Yamamoto, D., Ishiguro, S., Tomita, M., and Yachie, N. (2019) Fast and global detection of periodic sequence repeats in large genomic resources. Nucleic Acids Res. Jan 25 2019. 47(2):e8. doi: 10.1093/nar/gky890.
近年、CRISPR-Cas9などのゲノム編集技術の開発が進み、様々な生物種において任意の染色体DNA配列を自在に書き換えることが可能になりつつある。これらの技術は生物学を大きく推進することはもちろん、医療分野、産業分野への応用開発も進んでいる。一方、世界中でこれらの技術とこれらに関連した知財を巡る争いも熾烈で、高性能で新たなゲノム編集技術を開発することも重要な課題である。
これまで開発されてきたゲノム編集技術に関連するタンパク質や核酸は、周期的反復構造をもつという特徴がある。それらは反復構造内の可変アミノ酸残基(または塩基配列)の組み合わせで特定の塩基対を認識している。例えば、Zinc finger(ZNF)やTranscriptional activator-like effector(TALE)は反復構造を持つアミノ酸配列を有する。さらに、近年急速に利用が広がっているCRISPR-Cas9も、元来はバクテリアのプラスミドやウィルスなどの外来から侵入してくるDNA配列に対する免疫獲得機構として発見され、免疫の際に、リピート配列間の領域に一定の長さの外来DNA配列が保存されていくことで周期性が生じる。加えて、反復配列はゲノムの進化に深く関連していることが知られており、原核生物では反復単位の収縮、拡張によって遺伝子の発現や機能を変化させることで、環境変化や宿主からの免疫に応答する相変異(Phase variation)という仕組みを持っている。その他にもTetratricopeptideリピート(TPR)、Ankyrinリピート(ANK)、WD40リピート等の大規模で周期的な反復タンパク質ファミリーは、タンパク質間相互作用を仲介し、遺伝子発現、細胞周期、シグナル伝達等の様々な生物学的プロセスの制御に関連している。このように、周期的な反復配列を探索することは、生物学的に重要な発見及び、新規ゲノム編集ツール候補の発見・開発につながる可能性があるにもかかわらず、これまでゲノム・メタゲノムリソースからの周期性を有する反復配列をその種類に関係なくde novoかつ高スループットに検出するソフトウェアは現状まだ少ない。
今回、慶應義塾大学大学院政策・メディア研究科後期博士課程1年(当時)の森秀人氏らは、k-merを用いた反復性及び周期性評価に基づいて大規模なゲノムリソースから網羅的に周期的リピート配列をde novoに検出することができるソフトウェアSPADE(Search for Patterned DNA Elements)を開発した。筆者らは実際に18のTALE遺伝子領域と1つのCRISPR領域を有するXanthomonas oryzaeのゲノムをSPADEで解析したところ、それら既知の周期的配列をアノテーション通りに全て検出した(図a、b)。また、7,006の原核生物ゲノムを対象に、CRISPR領域の予測に広く用いられているソフトウェアのCRISPRFinderと検出精度をベンチマークセットに対し比較した。その結果、SPADEとCRISPRFinderはそれぞれを99.5%及び98.1%捕捉し、同等以上の検出精度があることを示した。さらに、タンパク質において反復モチーフ配列に揺らぎがある変性リピートのデータセットについても、タンパク質性リピートの検出用ソフトウェアであるXSTREAM、T―REKSとの検出精度の比較を行った。SPADEはデータセットに含まれる各タンパク質ファミリーの全てをこれらのソフトウェア間で最も良く検出すると同時に、その反復モチーフに揺らぎがある変性リピートに対しても、その周期を正確に決定できることが示された(図c)。加えて、SPADEによって検出された新規ゲノム編集関連遺伝子の候補には、近年植物から発見され国産ゲノム編集技術としての応用が期待されるPPR様の遺伝子も存在し、さらには微生物ゲノム進化の推進に関わったと考えられる周期的リピート配列等も確認された。
現在、森氏らは新たな新規ゲノム編集関連遺伝子候補群の検証実験を進めるとともに、SPADEをもちいた解析対象を、真核生物ゲノムやメタゲノムといった対象に広げている。「今後、現在利用されているゲノム編集ツールを超えるような新たなゲノム編集ツールの開発や、このような自然界にありながら人工的にも見える規則的な配列から、全く新しい生物学的発見をしたい」と森氏は語った。
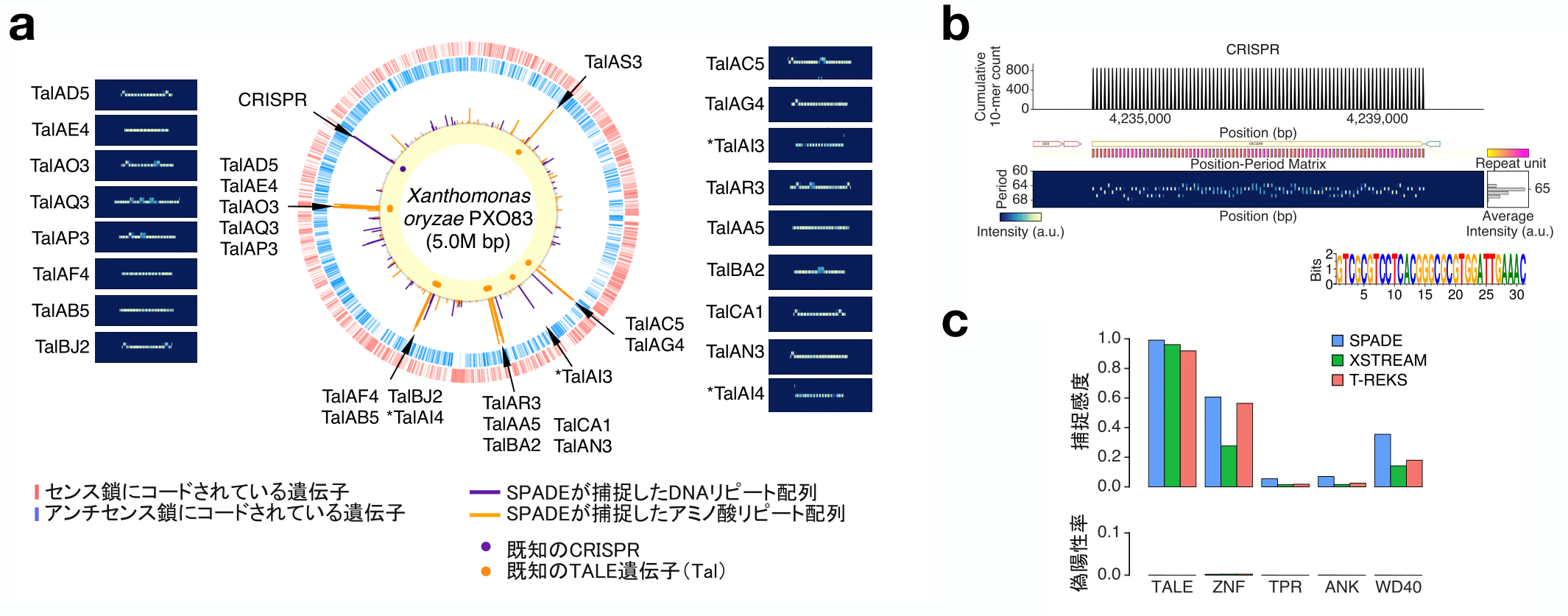
図a SPADEの解析例。Xanthomonas oryzaeゲノムにおいて全てのTALE遺伝子群とCRISPR領域が捕捉された。
図b Xanthomonas oryzaeゲノム中でSPADEが捕捉したCRISPR領域。正確にその領域、リピート周波数、繰返しモチーフが捉えられている。
図c 周期的アミノ酸配列を捕捉するソフトウェア群とSPADEを比較した例。様々な既知のリピート構造をSPADEが最も高感度に捉えた。
[編集:山本 楠]



