HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  ヒト免疫不全ウイルス (HIV) 進化の軌跡を俯瞰する
ヒト免疫不全ウイルス (HIV) 進化の軌跡を俯瞰する
19.06.27
ヒト免疫不全ウイルス (HIV) 進化の軌跡を俯瞰する
(19.06.27)
配列類似性ネットワークによるヒト免疫不全ウイルス(HIV) の分子進化解析
Nagata, S., Imai, J., Makino, G., Tomita, M., and Kanai, A. (2017). Evolutionary analysis of HIV-1 Pol proteins reveals representative residues for viral subtype differentiation. Front. Microbiol.8:2151. doi:10.3389/FMICB.2017.02151.
PMID: 29163435
1型ヒト免疫不全ウイルス (HIV-1)は、後天性免疫不全症候群(AIDS)の原因ウイルスであり、アフリカで祖先ウイルスがチンパンジーからヒトに種間感染した後、世界中へ感染が拡大した。HIV-1は高い進化速度と遺伝的多様性を持ち、ゲノム配列の違いにより複数の系統に分類されている。世界で主に流行している系統はグループMと呼ばれ、さらに細かな系統関係の違いにより9種類のサブタイプに分類されている。また、HIV-1のサブタイプによって病気の進行率や感染率に違いがあることが確認されている。その一方で、どのような機能を持つ領域がサブタイプの分化と対応して変化しているか、の全体像は、ほとんど明らかにされていなかった。
HIV-1のサブタイプは、これまで主にコア遺伝子 (gag、pol、env)の塩基やアミノ酸配列で系統樹を構築して、その系統関係を見ることにより分類されてきた。系統樹として表すことで各々の配列の関係を二分枝のモデルとして把握することができる一方、HIV-1で頻繁に発生するような組換え体を含む配列では、適切な系統樹を構築するには限界があった。そこで、慶應義塾大学環境情報学部(当時)の永田祥平氏らは、まず各Polタンパク質配列間の類似度に基づいて重み付き無向グラフ (配列類似性ネットワーク)を構築して描画することで、HIV-1サブタイプ全体の類似関係を可視化して俯瞰した。次にPolタンパク質上の機能性ドメイン領域配列を抽出し、ドメインごとの配列類似性ネットワークを構築して比較することで、サブタイプの違いを特徴付ける機能性領域が逆転写酵素コネクションドメインとRNase H領域と呼ばれるドメインであることを突き止めた(図A)。さらにこれら領域の中でどのアミノ酸残基がサブタイプの違いを特徴付けているのか明らかにするため、アライメントサイトごとにアミノ酸出現頻度の違いを分析した。その結果、得られたアミノ酸出現のパターンが大きく異なる残基はウイルス酵素の立体構造上で比較的近接して位置しており、また酵素の表面に存在することが明らかとなった(図B)。
さて、逆転写酵素コネクションドメインは逆転写酵素において結合溝を形成し、N末端ポリメラーゼ活性領域とC末端RNase H領域を結んでいる領域であり、RNase Hはウイルスゲノム複製時にDNA/RNA複合鎖のRNA鎖を特異的に分解する酵素である。逆転写酵素コネクションドメインとRNase H領域の2領域における変異が抗HIV-1薬(逆転写酵素阻害剤)に対する感受性を変化させていることが知られており、これは本研究で明らかになったPolタンパク質上でサブタイプを特徴付ける領域とドメインレベルで一致している。さらに、アミノ酸レベルで薬剤耐性変異残基と比較してみると、これら変異箇所とサブタイプ分化に対応している領域は完全に一致することはなく、お互いが非常に近傍に存在していた。薬剤耐性に強く関わる変異は逆転写酵素の活性との関連が深く、この位置への変異は逆転写酵素の構造を大きく変えてしまってその活性を下げてしまう可能性がある。そこでHIV-1は、これらの位置と非常に近傍に位置しているものの、配列保存性が低く、活性へ与える影響が小さい領域に変異を蓄積させて酵素機能をうまく最適化していきながらサブタイプを分化させていったものと推察される。
近年、感染が急拡大して問題となったエボラウイルスやジカウイルス等のエマージング・ウイルスも多くがHIV-1と同じRNAウイルスの一種であり、高い変異速度が特徴となっている。既にこれらウイルスのゲノム解析によって、感染が拡大していくと同時に配列上にも変異が蓄積していき、流行地域ごとの違いが生まれていることが明らかになっている。永田氏は「ウイルスが進化していく際に、ウイルスタンパク質の構造や機能にとって非常に重要な領域そのものではなく、その近傍を変化させるという考えは、これから新たに現れると考えられるエマージング・ウイルスの進化とそのサブタイプ分化に対しても適用できるだろう。」と語った。
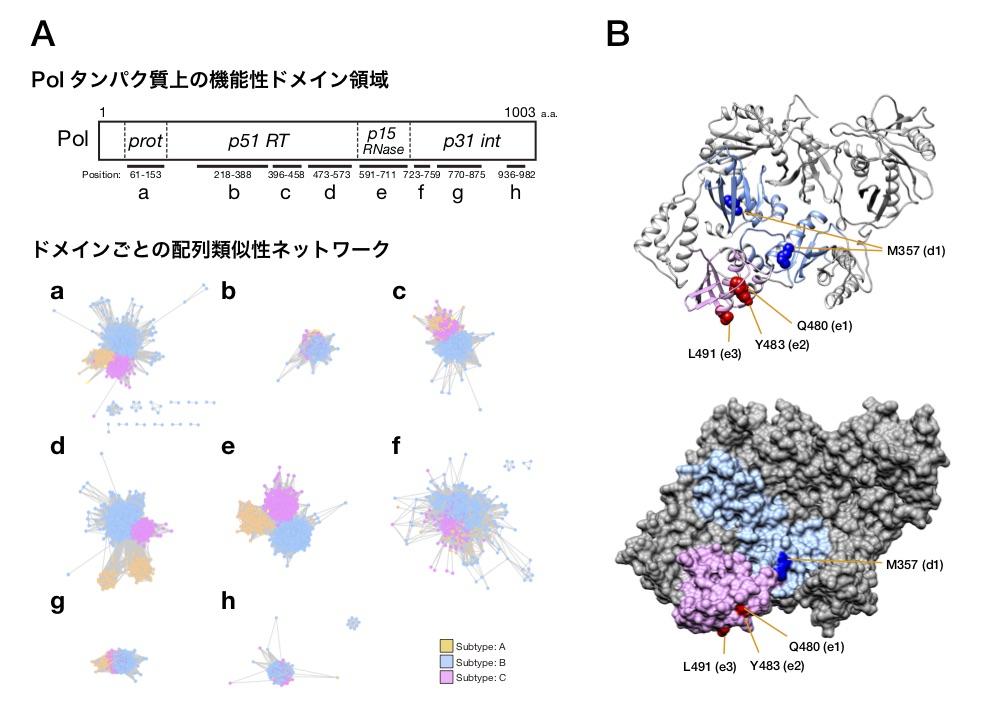
図:(A) HIV-1 Polタンパク質上の機能ドメイン領域ごとの配列類似性ネットワークの比較。a-hが各ドメインを表しており、dが逆転写酵素コネクションドメイン、eがRNase H領域である。
(B) サブタイプを特徴付ける領域の逆転写酵素立体構造上へのマッピング。水色領域が逆転写酵素コネクションドメインで、桃色領域がRNase Hを表している。アミノ酸レベルで分析した結果を濃く着色している。
[編集:山本楠]



