HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  tRNAリン酸化酵素の多様性はいかにして生み出されたのか、大規模分子進化解析でtRNAリン酸化酵素Clp1ファミリーの進化系譜が明らかに
tRNAリン酸化酵素の多様性はいかにして生み出されたのか、大規模分子進化解析でtRNAリン酸化酵素Clp1ファミリーの進化系譜が明らかに
20.04.18
tRNAリン酸化酵素の多様性はいかにして生み出されたのか、大規模分子進化解析でtRNAリン酸化酵素Clp1ファミリーの進化系譜が明らかに
(20.04.18)
tRNAリン酸化酵素の多様性はいかにして生み出されたのか
大規模分子進化解析でtRNAリン酸化酵素Clp1ファミリーの進化系譜が明らかに
Saito, M., Sato, A., Nagata, S., Tamaki, S., Tomita, M., Suzuki, H., and Kanai, A. (2019) Large-scale Molecular Evolutionary Analysis Uncovers a Variety of Polynucleotide Kinase Clp1 Family Proteins in the Three Domains of Life. Genome Biol Evol. 2019 Oct 1;11(10):2713-2726. doi: 10.1093/gbe/evz195.
そこで、慶應義塾大学大学院政策・メディア研究科の齋藤元文氏と金井昭夫教授らは、生物の3ドメインをまたぐ大規模な分子進化解析によって、Clp1タンパク質の系統学的分布を詳細に明らかにすることを目指した。より大規模な解析に向けて、齋藤氏らは複数のClp1タンパク質の酵素活性を担うPNK (Polynucleotide Kinase) ドメインを基に真核生物、古細菌、真正細菌についてClp1タンパク質の有無を探索した。その結果、Clp1遺伝子は真核生物1,426種、古細菌211種で同定されただけでなく、新たに真正細菌144種にも存在することが明らかになった。また、生物の3ドメインにおいて、共通のタンパク質ドメイン以外に差があることから、タンパク質ドメインの進化が起きたことが考えられた(図2)。興味深いことに、真正細菌のClp1は様々な門に広く分布している一方で、真正細菌全体の種数に対するClp1の存在比が極めて少なく、その多くが好熱環境に生育していた。次に齋藤氏らは、情報学的解析によって予測された新規真正細菌のClp1が、真核生物や古細菌のClp1と同様に核酸へのリン酸化機能を持つかを実験的に検証した。予測された真正細菌のClp1を大腸菌で組換え体タンパク質として精製し、DNAやRNAへのリン酸化機能を検証した結果、真正細菌Clp1はDNAとRNA両者をリン酸化し、特にRNAをよりリン酸化する傾向があることが明らかとなった。真核生物のClp1はRNAのみをリン酸化することから、真核生物のClp1と比較すると真正細菌のClp1は基質特異性が緩いと考えられる。さらに、真正細菌のClp1は90˚CでもRNAリン酸化活性を示す耐熱性タンパク質であった。この結果は、真正細菌のClp1が好熱環境に生息する古細菌由来の遺伝子であることを示唆している.今後、Clp1の構造的な特徴と酵素活性の関係性が明らかになれば、Clp1の関与が知られているヒトの神経病について新たな知見を得ることにも繋がると考えられる。
齋藤氏は「これからの生命科学の分野では、より膨大で複雑なデータを扱う必要が出てくると予想されます。その中で、意味のある情報を絞り込んだうえで実験学的に検証する、といったプロセスを如何に行うのか。この点は、本研究のような分子進化学のみならず、複雑な生命現象を理解していく上で重要であると思いますし、今後も多角的なアプローチによって研究進めていきたいです。」と語った。
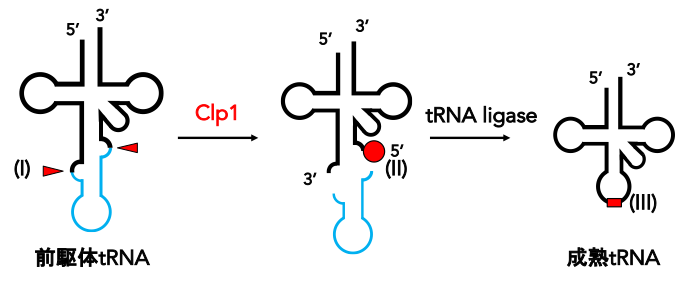
図1. 前駆体tRNAから成熟tRNAが合成される一過程
(I) 前駆体tRNA のイントロン(水色)がスプライシング酵素によって除去され,(II) tRNA のエクソン(黒色)の5'末端がClp1によってリン酸化を受け(赤丸),(III) tRNA ligaseによってtRNAエクソン(黒色)同士が連結されることで,機能的な成熟tRNAが合成される.
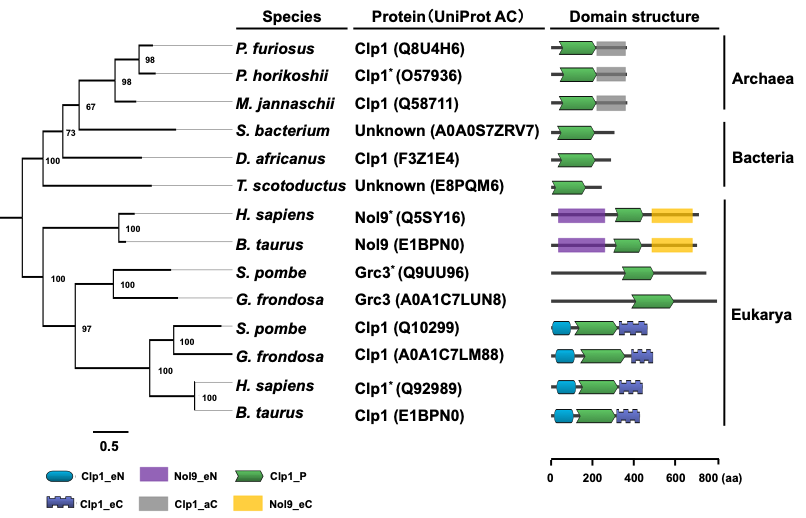
図2. 真核生物,古細菌,真正細菌Clp1を用いた分子進化系統樹
Clp1は真核生物 (Eukarya),古細菌 (Archaea),真正細菌 (Bacteria)に広く分布し,リン酸化に必須のClp1_Pドメイン(緑色)はすべての生物種のClp1で保存されていた.
[編集:河野夏鈴]



