HOME  論文/ハイライト
論文/ハイライト  研究ハイライト
研究ハイライト  論文ハイライト
論文ハイライト  遺伝子発現データから乳がん転移を予測する手法を開発
遺伝子発現データから乳がん転移を予測する手法を開発
20.07.31
遺伝子発現データから乳がん転移を予測する手法を開発
(20.07.31)
変数選択によって汎化性能を大幅に向上
Hikichi S, Sugimoto M. and Tomita M. (2020) Correlation-centred variable selection of a gene expression signature to predict breast cancer metastasis. Sci Rep. 10, 7923. doi: 10.1038/s41598-020-64870-z.
乳がんは世界中の女性の間で最も一般的ながんである。がんの治療効果や予後の予測を行う研究として、数万個の遺伝子のmRNAを同時に測定するマイクロアレイデータなどを使用し、複数の遺伝子の発現情報の組み合わせを用いるものがこれまでに数多く報告されてきた。乳がんでは、手術によって切除された腫瘍組織の一部を用いて70遺伝子の発現量から乳がんの再発リスクを予測する"MammaPrint"や、がん組織の21遺伝子の発現を調べることで化学療法の効果や予後を分析する"Oncotype DX"などの商用の検査に活用されている。これらの検査では多数の遺伝子を使うものが多いが、要素数が多いと限定された条件の精度が高くなるものの、その条件に特化した学習をしてしまうため、条件が変わった場合の予測性能(汎化性能)が低くなる傾向がある。よって、できるだけ少ない遺伝子で高精度・高汎用性で予測することが望まれる。
そこで、慶應義塾大学大学院政策・メディア研究科博士課程、日本学術振興会特別研究員(当時)の引地志織氏らは、高い予測能力と汎化能力の両方を備えた最小限の遺伝子セットを選択するために、分散拡大係数(VIF)を利用した独自の方法で遺伝子発現データの解析に取り組んだ。まず、乳がん組織のマイクロアレイデータを利用し、リンパ節陰性乳がんの患者の転移で変動のある遺伝子をt検定により抽出後、変動量によってランク化し、上位のものをサポートベクターマシンによる再帰的特徴消去(SVM--RFE)によって選択した。続いて、これら候補を分散拡大係数(VIF)によって独立な最小セットに絞り込み、特徴遺伝子を抽出した。さらに、これらの遺伝子数において汎化性能を目的関数として交差検証によって絞り込み、最終的に12遺伝子に落とし込んだ。先行研究(Wang et al ., 2005)は同じデータ(22283遺伝子)を用いて、多重ロジスティック回帰によって76遺伝子を抽出したことを考えると、大幅に絞り込みが加えられていることがわかる。Wangらの手法は学習データにおいては高い予測性能を持つものの、検証データにおいては12遺伝子を用いた本研究の手法が有意に予測性能において上回った。同様に、異なるデータセットを用いた検証においても、本研究の学習手法の方が、予測性能が高く、汎化性能の向上が示された。一方、どのような遺伝子が選択されたのかを遺伝子の機能分類であるGene Ontologyを用いて解析したところ、Wangらの学習結果のうち、「オルガネラ構築」に関わる遺伝子が選択的に濃縮された結果がこの12遺伝子であり、生物機能的にも合理的な遺伝子が抽出されてきた可能性がある。引地氏らが開発した手法により、既存の商用検査であるMammaPrintやOncotype DXで利用されている遺伝子よりも少ない変数で、高精度・高汎用性で5年以内の転移を予測することが可能となった。これは、新たに得られたデータの特徴選択を行う際の実用性を考える上で重要な特徴であり、引地氏らが提案するアプローチは乳がん患者の治療に貢献できると期待される。
引地氏は「本研究はがん診断における転移を予測するバイオマーカー探索の研究として有用であると考えており、今後も精力的に研究に取り組んでいきたいです。また、生命科学(分子生物学,バイオインフォマティクス)と情報工学(Semantic Computing,Data Mining)の両観点からの研究経験を生かし、個別化医療に関する医学・生命科学データの分析手法を提案できる研究者を目指します。」と語った。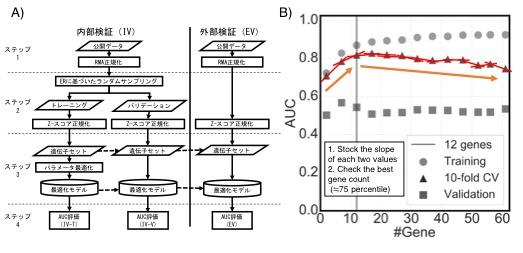
図: リンパ節陰性乳がんの5年以内の転移を予測する遺伝子選択方式(相関中心変数選択方法)
A) 遺伝子セット同定のためのフローチャート、B) 遺伝子数に着目した3つのデータセットの予測性能と最適遺伝子数決定方法
[編集: 安在麻貴子]



